Informationsbehandling är viktigt inom alla vetenskapsområden. I molekylärbiologi är den centrala dogmen, som först myntades av Francis Crick (Crick, 1958, 1970), en klassisk ryggrad i levande celler för att i grunden utföra processer från celldelning till död genom DNA-, RNA- och proteininformationsvägarna. Mer specifikt beskriver det centrala dogmet överföringen av sekvensinformation under DNA-replikering, transkription till RNA och translation till aminosyrakedjor som bildar proteiner. Samtidigt står det också att information inte kan flöda från protein till protein eller nukleinsyra.
Sedan systemiska och höga genomströmningsmetoder har kommit under de senaste två decennierna, har dessa breda steg, som inte inkluderar komplexa regleringsuppgifter har granskats intensivt. De saknade reglerande funktionerna, såsom DNA-korrekturläsning / reparationsmekanismer och alternativ skarvning av pre-mRNA, introducerar flera mellanliggande steg. Dessa ytterligare steg stör dogmens nyckelsteg och förändrar sannolikt informationsdynamiken. Dessutom verkar epigenetik, eller den roll som kromatinstrukturer, DNA-metylering och histonmodifikationer spelar, strida mot dogmens enkla vägar (Shapiro, 2009; Luco et al., 2011). Proteinsplitsning, eller förmågan hos ett protein (intein) att ändra sin egen sekvens, upptäckt på senare tid (Volkmann och Mootz, 2012) och prioner, som modifierar andra proteinsekvenser (Prusiner, 1998), kringgår informationsöverföringsvägen för dogm. Andra undersökningar rapporterade fel eller felaktiga matchningar mellan RNA-sekvenser och deras kodande DNA (Hayden, 2011; Li et al., 2011). Sammantaget ifrågasätter dessa data giltigheten av det centrala dogmet i dagens vetenskap och ifrågasätter därför enkelheten i linjärt informationsflöde (DNA till RNA och RNA till protein).
För att sätta saker i perspektiv kräver vi analytiska verktyg som undersöker oro eller avvikelser angående den långvariga teorin. En enkel, men ändå mycket användbar teknik för att söka globala egenskaper i datamängder med hög kapacitet är statistisk korrelationsanalys, som har använts i stor utsträckning och framgångsrikt för att observera mönster i komplexa system som väder (Stewart, 1990), aktiemarknader (Lo och MacKinlay , 1988) och kosmologi (Amati et al., 2008). Det finns flera typer av korrelationsanalyser som utvärderar både linjära (t.ex. Pearson produktmoment) och icke-linjära (t.ex. Spearmans rang, ömsesidig information) beroende (Steuer et al., 2002; Rosner, 2011). har korrelationsanalysen från Pearson produktmoment blivit den mest populära på grund av dess förmåga att visa organisationsstruktur i den enklaste formen.
I biologin har det funnits ett flertal verk som har studerat korrelationerna i mRNA och proteinuttrycksdata (se nedan och tabell 1). I teorin, när två prover som innehåller högdimensionella (såsom mikroarray och proteomiska) data jämförs, ger korrelationsanalyserna ett mått på avvikelse från enhet som en skillnadskälla mellan proverna Kortfattat visar två prover med identisk och fullständigt icke-identisk information enhet (R2 = 1) respektive null (R2 = 0) korrelation.
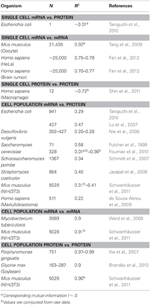
Tabell 1. mRNA och protein expr essionskorrelationer i olika organismer.
Perfekt korrelation (R2 = 1) är en idealiserad situation som är långt ifrån verkligheten, som teknisk eller experimentellt buller enbart stör och minskar korrelationen. Dessutom har de senaste åren belyst förekomsten av biologiskt brus: studierna på enskilda celler och molekyler har visat stokasticitet i genuttrycksdynamik på grund av den kombinatoriska effekten av låga molekylära kopieringsantal och den kvantala naturen hos promotordynamiken (Raj och van Oudenaarden, 2009; Eldar och Elowitz, 2010). Å andra sidan uppvisar klonala populationer av celler heterogenitet i nivåerna av ett givet proteinuttryck per cell vid varje uppmätt tidpunkt (Chang et al., 2008). Tillsammans är stokasticitet och heterogenitet väsentliga för att producera celldödsdiversifiering, fenotypiska variationer och förstärkning av intracellulära signaler (Locke et al., 2011; Selvarajoo, 2012).
De stokastiska fluktuationerna eller inneboende buller orsakar uttrycket av en molekylär art att variera i tid och mellan celler, vilket leder till okorrelerade svar (Elowitz et al., 2002). Detta är särskilt framträdande för mRNA och proteiner med låga kopieringsantal. Således kan korrelationen mellan prover (celler) sänkas på grund av inneboende brus (figur 1A). Andra källor till biologiskt buller på grund av yttre faktorer inkluderar variation i cellstorlek, antal molekylära kopior och fluktuationer i omgivningen mellan enskilda celler.Dessa faktorer snedvrider det deterministiska centrala dogmet och förändrar sannolikt starka korrelationer till svagare (Figur 1B).
En nyligen genomförd studie jämförde Escherichia coli mRNA och proteinuttryck mellan enskilda celler på enstaka molekylnivå och gav ett scenario som djupt ifrågasätter det centrala dogmet. Taniguchi et al. (2010) avslöjade att det inte finns någon korrelation (R2 ~ 0) mellan enskilda tufA-mRNA och proteinnivåer i enstaka celler. Särskilt drog de slutsatsen att bristen på korrelation sannolikt beror på skillnader i mRNA och proteinlivstid. Även om detta är en rimlig förklaring, Taniguchi et al. var noga med att inte motbevisa den långvariga hypotesen genom att hävda att tidsgenomsnitt av mRNA-nivåer skulle korrelera med proteinnivåer. Det fanns dock inga bevis som visade att detta är det faktiska fallet, och när vi utvärderade icke-linjära beroenden med ömsesidig information (Steuer et al., 2002; Tsuchiya et al., 2010) i Taniguchi et al. datamängden, fann vi att resultatet inte var beroende, dvs I ~ 0. Detta bekräftar att mRNA till proteinuttryck mellan enskilda celler på enskild molekylnivå är uppenbart orelaterade. Vidare, när man zoomar på enstaka molekylnivå i korrelationsdiagrammet, är det uppenbart att deras parvisa korrelationer är svaga (figur 1A, infoga, för illustration).
Särskilt på cellpopulationsnivå, Taniguchi et al. kunde visa relativt hög korrelation mellan mRNA och proteinuttryck med R2 = 0,29 (figur 2A). Faktum är att en annan oberoende studie av Lu et al. (2007), för E. coli-populationen, visade också relativt hög korrelation (R2 = 0,47). Liknande analyser utförda på Saccharomyces cerevisiae (Futcher et al., 1999), murin NIH / 3T3 fibroblast (Schwanhäusser et al., 2011) och flera andra cellpopulationer (Nie et al., 2006; Schmidt et al., 2007; Jayapal et. al., 2008; de Sousa Abreu et al., 2009) visade alla korrelerade strukturer mellan transkriptomomfattande och proteomomfattande uttryck (tabell 1). Så varför finns det ingen korrelation mellan enskilda mRNA och proteinuttryck i enstaka celler, medan på populationsnivå observeras kollektiva relationer mellan storskaligt mRNA och proteinuttryck?
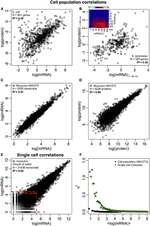
Figur 2. Korrelationer mellan uttrycksomfattande uttryck. Cellpopulationer: mRNA-proteinkorrelationer i (A) E. coli (Taniguchi et al., 2010) och (B) S. cerevisiae (Fournier et al., 2010) mellan mRNA-uttryck vid t = 60 min och proteinuttryck vid t = 360 min. Infoga: korrelationsmatris mellan alla tidpunkter visar en fördröjd ökning av korrelationer mellan mRNA och proteiner. (C) mRNA och (D) proteinuttryck mellan två prover av murina NIH / 3T3-celler (Schwanhäusser et al., 2011). Enstaka celler: (E) mRNA-uttryck mellan två äggceller (Tang et al., 2009). De röda prickade linjerna anger regionerna med låga mRNA-uttryck (log (mRNA) < 5). (F) Buller (η2) kontra log (mRNA-uttryck) för cellpopulation (NIH / 3T3, svarta prickar, Schwanhäusser et al., 2011) och enstaka celler (Oocyter, gröna trianglar, Tang et al., 2009). Varje punkt representerar värdet för en grupp av P = 100 mRNA. η2 är nära noll för cellpopulationen för alla mRNA-uttryck. För enstaka celler är η2 högst för mRNA med lägsta kopieringsnummer och närmar sig noll för högre kopieringsnummer.
Vi tror det finns två huvudorsaker till skillnaderna. För det första, som tidigare nämnts, minskar buller, oavsett om det är biologiskt eller icke-biologiskt till sin natur, korrelationen. Eftersom analyser på enstaka celler har visat vikten av stokasticitet och variation, är dessa effekter avgörande för att minska enstaka cellkorrelationer. På ensemblenivå, när celler samplas till en population, minskas det totala (inneboende + yttre) bruset, eftersom slumpmässigt brus avbryts över alla spektrum av molekylära uttryck (figur 1C – F) för att avslöja genomsnittligt svar och självorganisation ( Karsenti, 2008; Selvarajoo, 2011; Hekstra och Leibler, 2012; Selvarajoo och Giuliani, 2012). Därför uppstår en bra grad av mRNA-proteinuttryckskorrelation. För det andra jämfördes individuell mRNA-proteinuttryckskorrelation mellan flera celler för enkelcellstudien (Taniguchi et al., 2010). I cellpopulationsstudier görs dock jämförelsen i sin helhet, över tusentals mRNA och proteiner över flera storleksordningar större än det uttrycksintervall som finns för en enda molekyl mellan celler. Detta leder därför till högre korrelationer på populationsnivå då effekten av enstaka molekylära variationer blir försumbar.
Trots att korrelerade strukturer observeras för cellpopulationer finns det konkreta skäl till den stora avvikelsen från perfekt korrelation.Som nämnts tidigare är en viktig punkt att mRNA och proteiner är sekventiellt belägna med flera saknade processer, ej representerade i det centrala dogmet. Att lägga till de saknade mellanprodukterna längs en biokemisk väg kommer att medföra en märkbar fördröjning i informationsflödet (Selvarajoo, 2006, 2011; Piras et al., 2011), och korrelationen mellan dem kan drabbas av detta. Detta kan också vara en del av det faktum som noterats av Taniguchi et al. att mRNA och proteinuttryck har olika livstid. Anmärkningsvärt stöds denna postulation i ett nyligen genomfört arbete med S. cerevisiae behandlat med Rapamycin som visade att de temporala korrelationerna av mRNA-proteinuttryck initialt var låga, R2 = 0,01 vid 40 minuter, dock över 360 minuter efter störning ökade korrelationen, R2 = 0,36 (Fournier et al., 2010, figur 2B). Uppgifterna indikerar att vid kemisk störning avviker det initiala svaret mellan mRNA och proteinuttryck på grund av tidsfördröjning och olika kinetiska mekanismer mellan dem, liksom sekundära effekter såsom autokrin eller parakrin signalstörning (Shvartsman et al., 2002; Isalan et al., 2008). När effekterna av störningen försvagas över tiden inträffade återhämtningen av korrelationer.
För att ytterligare kontrollera postulationen att sekventiella fördröjningsprocesser eller olika livstider är avgörande för att minska mRNA-proteinkorrelationer jämförde vi R2 mellan samma molekylära arter av det centrala dogmet (t.ex. mellan mRNA och mRNA) i cellpopulationer och enstaka celler. Den transkriptomövergripande mRNA-mRNA-expressionskorrelationen mellan replikat av NIH / 3T3 (Schwanhäusser et al., 2011) (figur 2C) och Mycobacterium tuberculosis (Ward et al., 2008) cellpopulationsprover är båda mycket höga, med R2 > 0,9 (tabell 1). Sådana starka korrelationer observeras också mellan populationsprover för protein-proteinuttryck i NIH / 3T3-celler (Schwanhäusser et al., 2011) (figur 2D), Porphyromonas gingivalis (Xia et al., 2007) och Glycine max (Brandão et al. , 2010) (Tabell 1). Eftersom dessa data som jämför samma art ger mycket höga korrelationer är det tänkbart att de sekventiella fördröjningsprocesserna eller olika livstider är ansvariga för att sänka korrelationsstrukturerna för populationsnivån mellan mRNA och proteinuttryck.
I enstaka murina äggceller ( Tang et al., 2009), när man jämför hela mRNA-mRNA-uttryck, observeras en starkt korrelerad struktur (R2 = 0,92, figur 2E). Med fokus endast på lågt uttryckta mRNA (med logaritmiska uttryck < 5) sänker det stokastiska bruset den parvisa korrelationen ganska dramatiskt (R2 < 0,54). För att undersöka detta resultat utvärderade vi brus, η2 = σ2XY / μ2XY, över hela mRNA-uttryck (figur 2F). Vi noterade att η2 är högst för de lägsta uttrycken på grund av den uttalade effekten av stokastiska fluktuationer jämfört med deras uttryck, och närmar sig noll för högre uttryck, där sådant brus blir mindre signifikant (Piras et al., 2012). För cellpopulation observeras, som förväntat, nära nollbrus över hela uttrycksområdet på grund av att slumpmässigt brus avbryts (figur 1E, F).
Högt korrelerade strukturer för hela mRNA – mRNA-uttryck var också rapporterad för enstaka cancerceller (Fan et al., 2012), om än mindre signifikant med R2 ~ 0,7 (tabell 1). Dessutom visade jämförelse mellan protein och proteinuttryck i LPS-stimulerade humana makrofager höga korrelationer, R2 ~ 0,72 (Shin et al., 2011) (Tabell 1). Även om det inte finns någon korrelation mellan enskilda mRNA-proteinuttryck i enstaka celler, är den storskaliga eller omiska hela korrelationen mellan samma molekylära arter i enstaka celler mycket hög.
Således, oavsett om enstaka celler eller cellpopulationer , indikerar omics-omfattande data att korrelationerna mellan samma molekylära arter (mRNA vs. mRNA och protein vs. protein) är märkbart högre än mellan olika arter (mRNA vs. protein). Detta speglar det faktum att även om tidsfördröjningsprocesser och olika livstider är nyckeln för att minska korrelationerna, är dessa mekanismer inte tillräckliga för att stödja bristen på korrelationsstruktur som observerats mellan enskilda cellers ”individuella transkript till proteinuttryck.
Så genom att undersöka storskaliga uttryck av mRNA och proteiner i olika cellulära system har vi visat att korrelationsstrukturer uppstår i global skala. Korrelationsanalyserna avslöjar dock endast anslutningen mellan två testade prover och visar inte riktningen För att det centrala dogmen ska vara giltigt i en global skala bör det totala informationsflödet vara från DNA till proteiner. Sådant informationsflöde har demonstrerats av otaliga andra studier som involverar störning av receptorer från cellpopulationer och övervakning av de resulterande dynamik av transkriptionsfaktorer som binder till DNA och induktion av storskaliga genuttryck (figur 3A).I fallet med LPS-stimulerade immunceller har det till exempel visats att aktiveringen av transkriptionsfaktorn NF-KB sker omkring 15 minuter (Liu et al., 1999), induktion av dess nedströmsgener vid cirka 30 minuter. min (Liu et al., 1999; Xaus et al., 2000; Selvarajoo et al., 2008) och översättning av motsvarande proteiner i området 60-90 min (Kawai et al., 1999; Xaus et al. ., 2000) (figur 3B). Sådan sekventiell riktning av den totala transkriptionen till informationsflöde för translation observeras också för bakteriesystem, såsom E. coli, på cellpopulationsnivå (Golding et al., 2005).

Figur 3. Informationsflödet för centrala dogmer. (A) Schematisk bild av LPS / TLR4-inducerad TNF-expression, via transkriptionsfaktor NF-KB och tnf-gen, efter linjärt informationsflöde. (B) Experimentella temporala profiler av promotorbindande aktivitet av NF-KB (övre paneler), tnf (mittpaneler) och TNF (nedre paneler) uttryck på cellpopulation. (C) Schematiska temporala profiler av promotordynamik, mRNA och proteinuttryck på encellsnivå (Raj och van Oudenaarden, 2009).
Alternativt avslöjar undersökningar vid enstaka cellupplösning slumpmässiga fluktuationer över det linjära informationsflödet: transkriptionsfaktorerna som är bindande till DNA-promotorregionerna är kvantala, vilket resulterar i sprickbeteende för mRNA-transkriptionen och inducerar därefter variation i protein-translationen, även mellan identiska celler (figur 3C) (Raj och van Oudenaarden, 2009; Eldar och Elowitz, 2010; Locke et al., 2011; Hekstra och Leibler, 2012; Selvarajoo, 2012). Som ett resultat, vid en viss tidpunkt, är det individuella molekylära svaret för enstaka celler ganska bullrigt jämfört med befolkningens genomsnittliga skala (Selvarajoo, 2011).
Slutsatser
Exemplen som visas i denna uppsats belyser skillnaderna i ordningen på korrelationsvärden som observerats mellan arter i den centrala dogmen över cellpopulationer och enstaka celler. De statistiska analyserna från cellpopulationer ger en bild av att expressionskorrelationen mellan samma molekylslag är mycket hög och mellan arter är måttligt hög. Även om enstaka cellkorrelationer mellan samma art är jämförbara med cellpopulationer, visade de en bredare spridning i sina uttrycksdiagram på grund av den uttalade effekten av biologiskt brus, särskilt för transkriptioner med låga kopieringsantal. I synnerhet blir de enstaka cellerna ”parvis korrelation noll för enskilda molekyler (Taniguchi et al., 2010). Faktum är att stokastiska fluktuationer och variation i molekylära uttryck är kända för att vara funktionella för att generera beslut om cellöden och tippa cellulära tillstånd (Losick och Desplan, 2008; Eldar och Elowitz, 2010; Kuwahara och Schwartz, 2012). Vi tror att de starka omics-stora korrelationerna uppstår som ett resultat av täta gen- och proteinreglerande nätverk över tusentals molekyler (Barabási och Oltvai, 2004; Karsenti , 2008) vilket resulterar i framväxande genomsnittliga svar. Analysera ett litet antal eller enskilda molekyler kan inte korrelationsstrukturen observeras.
Sammantaget är det tänkbart att betrakta informationsflödet av enskilt DNA till protein kommer att ifrågasätta den centrala dogmen eftersom svaret från varje molekyl vid en viss tidpunkt sannolikt inte kommer att korrelera. Men globalt antyder observationen av det genomsnittliga deterministiska svaret att genens jämvikt IC-information finns kvar längst till höger om vägarna. Därför bör det centrala dogmet ses som ett makroskopiskt cellulärt informationsflöde i en omics-omfattande skala och inte i en enda gen till proteinnivå. Som sådan tror vi att dess enkelhet kommer att fortsätta att förbli en av de mest inflytelserika teoretiska pelarna i levande system.
Uttalande om intressekonflikter
Författarna förklarar att forskningen genomfördes i frånvaron av kommersiella eller ekonomiska förhållanden som kan tolkas som en potentiell intressekonflikt.
Bekräftelser
Kentaro Hayashi tackas för kommentarer. Forskningsfonden i Tsuruoka city och Yamagata Prefecture uppskattas för deras stöd.
Crick, F. (1958). Om proteinsyntes. Symp. Soc. Exp. Biol. 12, 139–163.
Pubmed Abstract | Pubmed fulltext
Crick, F. (1970). Centrala dogmer av molekylärbiologi. Nature 227, 561-563.
Pubmed Abstract | Publicerad fulltext
Hayden, E. C. (2011). Bevis på förändrat RNA väcker debatt. Nature 473, 432.
Pubmed Abstract | Publicerad fulltext | CrossRef Fulltext
Hekstra, D. R. och Leibler, S. (2012). Beredskap och statistiska lagar i replikerade mikrobiella stängda ekosystem. Cell 149, 1164–1173.
Pubmed Abstract | Publicerad fulltext | CrossRef Fulltext
Kuwahara, H. och Schwartz, R. (2012).Stokastisk steady state-vinst i en genuttryckningsprocess med mRNA-nedbrytningskontroll. J. R. Soc. Gränssnitt 9, 1589–1598.
Pubmed Abstract | Publicerad fulltext | CrossRef Fulltext
Nie, L., Wu, G. och Zhang, W. (2006). Korrelation av mRNA-uttryck och proteinöverflöd som påverkas av flera sekvensfunktioner relaterade till translationell effektivitet i Desulfovibrio vulgaris: en kvantitativ analys. Genetik 174, 2229-2243.
Sammanfattning av Pubmed | Pubmed fulltext | CrossRef Fulltext
Prusiner, S. B. (1998). Prions. Proc. Natl. Acad. Sci. U.S.A. 95, 13363–13383.
Pubmed Abstract | Pubmed fulltext | CrossRef Fulltext
Rosner, B. (2011). Grunderna för biostatistik. 7: e Edn. Boston, MA: Duxbury Press.
Selvarajoo, K. (2006). Upptäcka differentiell aktiveringsmaskineri för vägtullsliknande receptor 4-signalvägar i MyD88-knockouts. FEBS Lett. 580, 1457–1464.
Pubmed Abstract | Pubmed fulltext | CrossRef Fulltext
Selvarajoo, K. (2011). Makroskopisk lag för bevarande avslöjad i populationsdynamiken för signalliknande receptorsignal. Cell Commun. Signal. 9, 9.
Pubmed Abstract | Publicerad fulltext | CrossRef Fulltext
Selvarajoo, K. (2012). Förstå multimodala biologiska beslut från enstaka celler och populationsdynamik. Wiley Interdiscip. Pastor Syst. Biol. Med. 4, 385–399.
Pubmed Abstract | Pubmed fulltext | CrossRef Fulltext
Stewart, T. R. (1990). En sönderdelning av korrelationskoefficienten och dess användning vid analys av prognosfärdigheter. Väderprognos. 5, 661–666.