O processamento da informação é essencial em todos os campos da ciência. Na biologia molecular, o dogma central, cunhado pela primeira vez por Francis Crick (Crick, 1958, 1970), é uma espinha dorsal clássica de células vivas para executar fundamentalmente processos desde a divisão celular até a morte através das vias de informação do DNA, RNA e proteínas. Mais especificamente, o dogma central descreve a transferência de informações de sequência durante a replicação do DNA, a transcrição em RNA e a tradução em cadeias de aminoácidos formadoras de proteínas. Ao mesmo tempo, ele também afirma que as informações não podem fluir da proteína para a proteína ou ácido nucleico.
Desde o advento das abordagens sistêmicas e de alto rendimento nas últimas duas décadas, essas grandes etapas, que não incluem detalhes regulatórios complexos estão sob intenso escrutínio. As características regulatórias ausentes, como os mecanismos de revisão / reparo de DNA e splicing alternativo de pré-mRNA, introduzem várias etapas intermediárias. Essas etapas adicionais interferem com as etapas principais do dogma e provavelmente alteram a dinâmica da informação. Além disso, a epigenética, ou o papel desempenhado pelas estruturas da cromatina, metilação do DNA e modificações nas histonas, também parecem ir contra as vias simples do dogma (Shapiro, 2009; Luco et al., 2011). O splicing de proteínas, ou a capacidade de uma proteína (inteínas) de alterar sua própria sequência, descoberta recentemente (Volkmann e Mootz, 2012) e os príons, que modificam outras sequências de proteínas (Prusiner, 1998), contornam a via de transferência de informações do dogma. Outras investigações relataram erros ou incompatibilidades entre as sequências de RNA e seu DNA codificador (Hayden, 2011; Li et al., 2011). Juntos, esses dados lançam dúvidas sobre a validade do dogma central no contexto da ciência atual e, portanto, questionam a simplicidade do fluxo linear de informações (DNA para RNA e RNA para proteína).
Para colocar as coisas em perspectiva, precisamos de ferramentas analíticas que investiguem as preocupações ou discrepâncias em relação à teoria de longa data. Uma técnica simples, mas altamente útil para pesquisar propriedades globais em conjuntos de dados de alto rendimento é a análise de correlação estatística, que tem sido amplamente e com sucesso usada para observar padrões em sistemas complexos como o clima (Stewart, 1990), mercados de ações (Lo e MacKinlay , 1988) e cosmologia (Amati et al., 2008). Existem vários tipos de análises de correlação que avaliam as dependências lineares (por exemplo, momento-produto de Pearson) e não lineares (por exemplo, classificação de Spearman, Informação mútua) (Steuer et al., 2002; Rosner, 2011). , a análise de correlação momento-produto da Pearson tornou-se a mais popular devido à sua capacidade de mostrar a estrutura organizacional da forma mais simples.
Em biologia, houve vários trabalhos que estudaram as correlações no mRNA e dados de expressão de proteína (ver abaixo e Tabela 1). Em teoria, quando duas amostras contendo dados de alta dimensão (como microarray e proteômica) são comparadas, as análises de correlação fornecem uma medida de desvio da unidade como uma fonte de diferença entre as amostras . Resumidamente, duas amostras com informações idênticas e completamente não idênticas mostrarão correlação unitária (R2 = 1) e nula (R2 = 0), respectivamente.
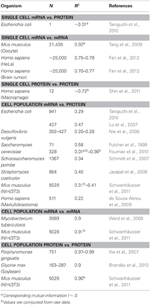
Tabela 1. mRNA e proteína expr correlações de essência em vários organismos.
Correlação perfeita (R2 = 1) é uma situação idealizada que está longe da realidade, como técnica ou o ruído experimental sozinho interfere e reduz a correlação. Além disso, os últimos anos destacaram a existência de ruído biológico: os estudos em células e moléculas individuais mostraram estocasticidade na dinâmica da expressão gênica devido ao efeito combinatório de baixos números de cópias moleculares e a natureza quantal da dinâmica do promotor (Raj e van Oudenaarden, 2009; Eldar e Elowitz, 2010). Por outro lado, as populações clonais de células apresentam heterogeneidade nos níveis de expressão de uma determinada proteína por célula em qualquer momento medido (Chang et al., 2008). Juntas, estocasticidade e heterogeneidade são essenciais para a produção de diversificação do destino celular, variações fenotípicas e amplificação de sinais intracelulares (Locke et al., 2011; Selvarajoo, 2012).
As flutuações estocásticas, ou ruído intrínseco, causam a expressão de uma espécie molecular para variar no tempo e entre as células, levando a respostas não correlacionadas (Elowitz et al., 2002). Isso é especialmente importante para mRNAs e proteínas com baixo número de cópias. Assim, a correlação entre amostras (células) pode ser reduzida devido ao ruído intrínseco (Figura 1A). Outras fontes de ruído biológico devido a fatores extrínsecos incluem variabilidade no tamanho da célula, número de cópias moleculares e flutuações ambientais entre células individuais.Esses fatores distorcem o dogma central determinístico e provavelmente alteram correlações fortes em mais fracas (Figura 1B).
Um estudo recente comparou o mRNA de Escherichia coli e as expressões de proteínas entre células individuais no nível de uma única molécula e forneceu um cenário que questiona profundamente o dogma central. Taniguchi et al. (2010) revelou que não há correlação (R2 ~ 0) entre o mRNA de tufA individual e os níveis de proteína em células individuais. Notavelmente, eles concluíram que a falta de correlação é provavelmente devido a diferenças no mRNA e no tempo de vida das proteínas. Embora essa seja uma explicação plausível, Taniguchi et al. tiveram o cuidado de não refutar a hipótese de longa data, alegando que as médias de tempo dos níveis de mRNA deveriam se correlacionar com os níveis de proteína. No entanto, não foram mostradas evidências para demonstrar que este é o caso real, e quando avaliamos as dependências não lineares usando informações mútuas (Steuer et al., 2002; Tsuchiya et al., 2010) em Taniguchi et al. conjunto de dados, descobrimos que o resultado não é dependente, ou seja, I ~ 0. Isso confirma que as expressões de mRNA para proteínas entre células individuais em nível de molécula única são claramente não relacionadas. Além disso, ao ampliar no nível de molécula única no gráfico de correlação, é evidente que suas correlações de pares são fracas (Figura 1A, inserção, para ilustração).
Notavelmente, no nível da população de células, Taniguchi et al. foram capazes de mostrar correlação relativamente alta entre as expressões de mRNA e proteínas com R2 = 0,29 (Figura 2A). Na verdade, outro estudo independente de Lu et al. (2007), para a população de E. coli, também apresentou correlação relativamente alta (R2 = 0,47). Análises semelhantes realizadas em Saccharomyces cerevisiae (Futcher et al., 1999), fibroblasto murino NIH / 3T3 (Schwanhäusser et al., 2011) e várias outras populações de células (Nie et al., 2006; Schmidt et al., 2007; Jayapal et al., 2008; de Sousa Abreu et al., 2009) todos mostraram estruturas correlacionadas entre as expressões do transcriptoma e do proteoma (Tabela 1). Então, por que não há correlação entre mRNA individual e expressões de proteínas em células individuais, enquanto no nível da população, relações coletivas são observadas entre mRNA em grande escala e expressões de proteínas?
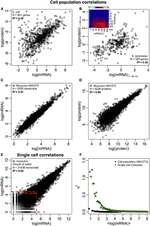
Figura 2. Correlações de expressão em todo o Omics. Populações celulares: correlações de mRNA-proteína em (A) E. coli (Taniguchi et al., 2010) e (B) S. cerevisiae (Fournier et al., 2010) entre expressões de mRNA em t = 60 min e expressões de proteína em t = 360 min. Inserção: a matriz de correlação entre todos os pontos de tempo mostra um aumento atrasado nas correlações entre mRNA e proteínas. (C) mRNA e (D) expressões de proteínas entre duas amostras de células NIH / 3T3 murinas (Schwanhäusser et al., 2011). Células individuais: (E) expressões de mRNA entre dois oócitos (Tang et al., 2009). As linhas vermelhas pontilhadas indicam as regiões de expressões de baixo mRNA (log (mRNA) < 5). (F) Ruído (η2) versus log (expressões de mRNA) para população de células (NIH / 3T3, pontos pretos, Schwanhäusser et al., 2011) e células únicas (Oócitos, triângulos verdes, Tang et al., 2009). Cada ponto representa o valor para um grupo de P = 100 mRNAs. η2 é quase zero para a população de células para todas as expressões de mRNA. Para células únicas, η2 é mais alto para mRNAs com o menor número de cópias e se aproxima de zero para maiores números de cópias.
Acreditamos existem duas razões principais para as diferenças. Em primeiro lugar, como observado anteriormente, o ruído, seja de natureza biológica ou não biológica, reduz a correlação. Uma vez que as análises em células individuais mostraram a importância da estocasticidade e variabilidade, esses efeitos são cruciais para reduzir as correlações de células únicas. No nível do conjunto, quando as células são amostradas em uma população, o ruído total (intrínseco + extrínseco) é reduzido, pois o ruído aleatório se cancela em toda a gama de expressões moleculares (Figuras 1C-F), para revelar a resposta média e auto-organização ( Karsenti, 2008; Selvarajoo, 2011; Hekstra e Leibler, 2012; Selvarajoo e Giuliani, 2012). Assim, surge um bom grau de correlação de expressão de mRNA-proteína. Em segundo lugar, para o estudo de célula única (Taniguchi et al., 2010), a correlação de expressão individual de mRNA-proteína foi comparada em várias células. Em estudos de população de células, no entanto, a comparação é feita na íntegra, em milhares de mRNAs e proteínas em várias ordens de magnitude maior do que a faixa de expressão encontrada para uma única molécula entre as células. Isso, portanto, leva a correlações mais altas no nível da população, pois o efeito de variações moleculares únicas torna-se insignificante.
Apesar de estruturas correlacionadas serem observadas para populações de células, há razões tangíveis para o grande desvio da correlação perfeita.Como observado anteriormente, um ponto-chave é que os mRNAs e as proteínas são sequencialmente localizados com vários processos ausentes, não representados no dogma central. Adicionar os intermediários ausentes ao longo de uma via bioquímica incorrerá em um atraso perceptível no fluxo de informações (Selvarajoo, 2006, 2011; Piras et al., 2011), e a correlação entre eles pode sofrer como resultado. Isso também pode ser parte do fato observado por Taniguchi et al. que as expressões de mRNA e proteína têm tempos de vida diferentes. Notavelmente, esta postulação é apoiada em um trabalho recente em S. cerevisiae tratado com Rapamicina que mostrou que as correlações temporais da expressão da proteína mRNA foram inicialmente baixas, R2 = 0,01 em 40 min, no entanto, mais de 360 min após a perturbação, a correlação aumentou, R2 = 0,36 (Fournier et al., 2010, Figura 2B). Os dados indicam que, mediante perturbação química, a resposta inicial entre as expressões do mRNA e da proteína se desvia devido ao atraso de tempo e diferentes mecanismos cinéticos entre eles, bem como efeitos secundários, como interferência de sinalização autócrina ou parácrina (Shvartsman et al., 2002; Isalan et al., 2008). Quando os efeitos da perturbação são atenuados ao longo do tempo, ocorre a recuperação das correlações.
Para verificar ainda mais a postulação de que os processos de atraso sequencial ou diferentes tempos de vida são cruciais para diminuir as correlações mRNA-proteína, comparamos R2 entre os mesmas espécies moleculares do dogma central (por exemplo, entre mRNA e mRNA) em populações de células e células individuais. A correlação de expressão de mRNA-mRNA de todo o transcriptoma entre réplicas de NIH / 3T3 (Schwanhäusser et al., 2011) (Figura 2C) e amostras de população de células de Mycobacterium tuberculosis (Ward et al., 2008) são ambas muito altas, com R2 > 0,9 (Tabela 1). Essas fortes correlações também são observadas entre amostras de população para expressões de proteína-proteína em células NIH / 3T3 (Schwanhäusser et al., 2011) (Figura 2D), Porphyromonas gingivalis (Xia et al., 2007) e Glycine max (Brandão et al. , 2010) (Tabela 1). Uma vez que esses dados que comparam as mesmas espécies geram correlações muito altas, é concebível que os processos de atraso sequencial ou diferentes tempos de vida sejam responsáveis por reduzir as estruturas de correlação do nível de população entre as expressões de mRNA e proteínas.
Em oócitos murinos únicos ( Tang et al., 2009), ao comparar expressões inteiras de mRNA-mRNA, uma estrutura altamente correlacionada é observada (R2 = 0,92, Figura 2E). No entanto, focando apenas em mRNAs expressos de forma humilde (com expressões logarítmicas < 5), o ruído estocástico reduz a correlação de pares de maneira bastante dramática (R2 < 0,54). Para testar esse resultado, avaliamos o ruído, η2 = σ2XY / μ2XY, em todas as expressões de mRNA (Figura 2F). Notamos que η2 é maior para as expressões mais baixas, devido ao efeito pronunciado das flutuações estocásticas em comparação às suas expressões, e se aproxima de zero para expressões mais altas, onde esse ruído torna-se menos significativo (Piras et al., 2012). Para a população de células, como esperado, ruído próximo de zero é observado em todo o intervalo de expressão devido ao cancelamento do ruído aleatório (Figuras 1E, F).
Estruturas altamente correlacionadas para expressões de mRNA-mRNA inteiras também foram relatado para uma única célula cancerosa (Fan et al., 2012), embora menos significativo com R2 ~ 0,7 (Tabela 1). Além disso, a comparação das expressões proteína-proteína em macrófagos humanos estimulados por LPS também mostrou altas correlações, R2 ~ 0,72 (Shin et al., 2011) (Tabela 1). Embora não haja correlação entre as expressões individuais da proteína mRNA em células individuais, a correlação em grande escala ou ampla ômica entre as mesmas espécies moleculares em células individuais é muito alta.
Assim, sejam células individuais ou populações de células , os dados ômicos indicam que as correlações entre as mesmas espécies moleculares (mRNA vs. mRNA e proteína vs. proteína) são visivelmente maiores do que entre diferentes espécies (mRNA vs. proteína). Isso reflete o fato de que, embora os processos de retardo de tempo e diferentes tempos de vida sejam essenciais para reduzir as correlações, esses mecanismos não são suficientes para suportar a falta de estrutura de correlação observada entre células individuais “transcrição individual para expressões de proteínas.
Então agora, através da investigação de expressões em larga escala de mRNAs e proteínas de vários sistemas celulares, mostramos que estruturas de correlação surgem em escala global. No entanto, as análises de correlação revelam apenas a conectividade entre duas amostras testadas, e não mostram a direção de fluxo de informações. Para que o dogma central seja válido em uma escala global, o fluxo geral de informações deve ser do DNA para as proteínas. Esse fluxo de informações foi demonstrado por uma miríade de outros estudos que envolvem perturbar os receptores das populações de células e monitorar o resultado dinâmica dos fatores de transcrição que se ligam ao DNA e a indução de expressões gênicas em grande escala (Figura 3A).Por exemplo, no caso de células imunes estimuladas por LPS, foi demonstrado que a ativação do fator de transcrição NF-κB ocorre por volta de 15 min (Liu et al., 1999), a indução de seus genes a jusante em cerca de 30 min (Liu et al., 1999; Xaus et al., 2000; Selvarajoo et al., 2008), e a tradução das proteínas correspondentes na região de 60-90 min (Kawai et al., 1999; Xaus et al. ., 2000) (Figura 3B). Essa direção sequencial da transcrição geral para o fluxo de informações de tradução também é observada para sistemas bacterianos, como E. coli, no nível da população de células (Golding et al., 2005).

Figura 3. O fluxo de informações do dogma central. (A) Esquema da expressão de TNF induzida por LPS / TLR4, via fator de transcrição NF-κB e gene tnf, seguindo fluxo de informação linear. (B) Perfis temporais experimentais de atividade de ligação do promotor de expressões de NF-κB (painéis superiores), tnf (painéis do meio) e TNF (painéis inferiores) ao nível da população de células. (C) Perfis temporais esquemáticos da dinâmica do promotor, mRNA e expressões de proteínas no nível de uma única célula (Raj e van Oudenaarden, 2009).
Alternativamente, as investigações em resolução de célula única revelam flutuações aleatórias sobre o fluxo de informação linear: a ligação dos fatores de transcrição às regiões promotoras de DNA é quantal, resultando no comportamento de explosão da transcrição do mRNA e, subsequentemente, induz variabilidade na tradução da proteína mesmo entre células idênticas (Figura 3C) (Raj e van Oudenaarden, 2009; Eldar e Elowitz, 2010; Locke et al., 2011; Hekstra e Leibler, 2012; Selvarajoo, 2012). Como resultado, em qualquer ponto de tempo específico, a resposta molecular individual para células individuais é bastante ruidosa em comparação com a escala média da população (Selvarajoo, 2011).
Conclusões
Os exemplos mostrados em este artigo destaca as diferenças na ordem dos valores de correlação observados entre as espécies no dogma central sobre populações de células e células individuais. As análises estatísticas das populações de células mostram que a correlação de expressão entre as mesmas espécies moleculares é muito alta e entre as espécies é moderadamente alta. Embora as correlações de uma única célula entre as mesmas espécies sejam comparáveis com as populações de células, elas mostraram uma maior dispersão em seus gráficos de expressão devido ao efeito pronunciado do ruído biológico, especialmente para transcritos com baixo número de cópias. Notavelmente, a “correlação de pares de células individuais torna-se zero para moléculas individuais (Taniguchi et al., 2010). Na verdade, as flutuações estocásticas e a variabilidade em expressões moleculares são conhecidas por serem funcionais na geração de decisão do destino celular e estados celulares de inclinação (Losick e Desplan, 2008; Eldar e Elowitz, 2010; Kuwahara e Schwartz, 2012). Acreditamos que as fortes correlações ômicas ocorrem como resultado de redes reguladoras de genes e proteínas estreitas em milhares de moléculas (Barabási e Oltvai, 2004; Karsenti , 2008) resultando em respostas médias emergentes. Analisando um pequeno número ou moléculas individuais, a estrutura de correlação não pode ser observada.
No geral, é concebível que a visualização do fluxo de informações de um único DNA para a proteína questionará o dogma central como a resposta de cada molécula em um único momento provavelmente não se correlacionará. No entanto, globalmente, a observação da resposta determinística média sugere que o equilíbrio líquido do geneto a informação ic permanece na extrema direita das vias. Portanto, o dogma central deve ser visto como um fluxo de informação celular macroscópica em uma escala ampla de ômicas, e não em um único gene para nível de proteína. Como tal, acreditamos que sua simplicidade continuará a ser um dos pilares teóricos mais influentes dos sistemas vivos.
Declaração de conflito de interesses
Os autores declaram que a pesquisa foi conduzida em a ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.
Agradecimentos
Agradecemos a Kentaro Hayashi pelos comentários. O fundo de pesquisa da cidade de Tsuruoka e Prefeitura de Yamagata é grato por seu apoio.
Crick, F. (1958). Na síntese de proteínas. Symp. Soc. Exp. Biol. 12, 139–163.
Resumo do Pubmed | Pubmed Full Text
Crick, F. (1970). Dogma central da biologia molecular. Nature 227, 561–563.
Pubmed Abstract | Pubmed Full Text
Hayden, E. C. (2011). A evidência de RNA alterado desperta o debate. Nature 473, 432.
Resumo do Pubmed | Texto completo do Pubmed | CrossRef Full Text
Hekstra, D. R. e Leibler, S. (2012). Leis de contingência e estatísticas em replicar ecossistemas microbianos fechados. Cell 149, 1164–1173.
Pubmed Abstract | Texto completo do Pubmed | CrossRef Full Text
Kuwahara, H. e Schwartz, R. (2012).Ganho de estado estacionário estocástico em um processo de expressão gênica com controle da degradação do mRNA. J. R. Soc. Interface 9, 1589–1598.
Resumo do Pubmed | Texto completo do Pubmed | CrossRef Full Text
Nie, L., Wu, G. e Zhang, W. (2006). Correlação da expressão de mRNA e abundância de proteínas afetadas por características de múltiplas sequências relacionadas à eficiência translacional em Desulfovibrio vulgaris: uma análise quantitativa. Genetics 174, 2229–2243.
Pubmed Abstract | Texto completo do Pubmed | CrossRef Full Text
Prusiner, S. B. (1998). Prions. Proc. Natl. Acad. Sci. EUA 95, 13363–13383.
Resumo Pubmed | Texto completo do Pubmed | CrossRef Full Text
Rosner, B. (2011). Fundamentos de Bioestatística. 7th Edn. Boston, MA: Duxbury Press.
Selvarajoo, K. (2006). Descobrir a maquinaria de ativação diferencial das vias de sinalização do receptor 4 toll-like em nocautes MyD88. FEBS Lett. 580, 1457–1464.
Resumo do Pubmed | Texto completo do Pubmed | CrossRef Full Text
Selvarajoo, K. (2011). Lei macroscópica de conservação revelada na dinâmica populacional da sinalização de receptores toll-like. Cell Commun. Sinal. 9, 9.
Resumo Pubmed | Texto completo do Pubmed | Texto completo CrossRef
Selvarajoo, K. (2012). Compreender as decisões biológicas multimodais de uma única célula e dinâmica populacional. Wiley Interdiscip. Rev. Syst. Biol. Med. 4, 385–399.
Resumo do Pubmed | Texto completo do Pubmed | CrossRef Full Text
Stewart, T. R. (1990). Uma decomposição do coeficiente de correlação e seu uso na análise da habilidade de previsão. Previsão do tempo. 5, 661-666.