정보 처리는 모든 과학 분야에서 필수적입니다. 분자 생물학에서 Francis Crick (Crick, 1958, 1970)에 의해 처음 만들어진 중심 교리는 DNA, RNA 및 단백질 정보 경로를 통해 세포 분열에서 죽음에 이르는 과정을 근본적으로 실행하는 살아있는 세포의 고전적인 백본입니다. 보다 구체적으로, 중심 도그마는 DNA 복제, RNA 로의 전사 및 단백질을 형성하는 아미노산 사슬로의 번역 동안 서열 정보의 전달을 설명합니다. 동시에 정보는 단백질에서 단백질 또는 핵산으로 이동할 수 없다고 명시합니다.
지난 20 년 동안 체계적이고 높은 처리량 접근 방식의 출현 이후 이러한 광범위한 단계는 다음을 포함하지 않습니다. 복잡한 규제 세부 사항은 철저한 조사를 받았습니다. DNA 교정 / 수리 메커니즘 및 pre-mRNA의 대체 스 플라이 싱과 같은 누락 된 규제 기능은 몇 가지 중개 단계를 도입합니다. 이러한 추가 단계는 교리의 주요 단계를 방해하고 정보 역학을 변경할 가능성이 있습니다. 또한 후성 유전학 또는 염색질 구조, DNA 메틸화 및 히스톤 변형이 수행하는 역할도 교리의 단순한 경로에 위배되는 것으로 보입니다 (Shapiro, 2009; Luco et al., 2011). 최근에 발견 된 단백질 스 플라이 싱 또는 단백질 (inteins)이 자신의 서열을 변경하는 능력 (Volkmann and Mootz, 2012)과 다른 단백질 서열을 수정하는 프리온 (Prusiner, 1998)은 정보 전달 경로를 우회합니다. 교조. 다른 조사에서는 RNA 서열과 코딩 DNA 사이의 오류 또는 불일치를보고했습니다 (Hayden, 2011; Li et al., 2011). 종합하면 이러한 데이터는 오늘날 과학의 맥락에서 중심 교리의 타당성에 의문을 던지고 따라서 선형 정보 흐름 (DNA에서 RNA로, RNA에서 단백질로)의 단순성에 의문을 제기합니다.
사물을 원근감있게 표현하려면 오랜 이론에 대한 우려 나 불일치를 조사하는 분석 도구가 필요합니다. 처리량이 많은 데이터 세트에서 글로벌 속성을 검색하는 데 간단하지만 매우 유용한 기술 중 하나는 통계적 상관 분석으로, 날씨 (Stewart, 1990), 주식 시장 (Lo 및 MacKinlay)과 같은 복잡한 시스템의 패턴을 관찰하는 데 널리 성공적으로 사용되었습니다. , 1988) 및 우주론 (Amati et al., 2008). 선형 (예 : Pearson product-moment) 및 비선형 (예 : Spearman s rank, Mutual Information) 종속성을 모두 평가하는 여러 종류의 상관 분석이 있습니다 (Steuer et al., 2002; Rosner, 2011). 특히 , Pearson product-moment 상관 관계 분석은 가장 단순한 형태로 조직 구조를 보여줄 수있는 능력으로 인해 가장 인기가 있습니다.
생물학에서는 mRNA와 상관 관계를 연구 한 많은 연구가있었습니다. 단백질 발현 데이터 (아래 및 표 1 참조) 이론적으로 고차원 (마이크로 어레이 및 프로 테오 믹) 데이터를 포함하는 두 개의 샘플을 비교할 때 상관 분석은 샘플 간의 차이의 원인으로 단일 성과의 편차 측정을 제공합니다. 간단히 말해, 정보가 동일하고 완전히 동일하지 않은 두 샘플은 각각 단위 (R2 = 1) 및 널 (R2 = 0) 상관 관계를 표시합니다.
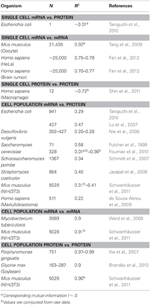
표 1. mRNA 및 단백질 expr 다양한 유기체에서의 ession 상관 관계.
완벽한 상관 관계 (R2 = 1)는 기술적으로 현실과는 거리가 먼 이상화 된 상황입니다. 또는 실험적 소음만으로도 상관 관계를 방해하고 감소시킵니다. 더욱이 최근 몇 년 동안 생물학적 소음의 존재를 강조했습니다. 개별 세포와 분자에 대한 연구는 저분자 복제 수의 조합 효과와 프로모터 역학의 양자 적 특성 (Raj 및 van Oudenaarden, 2009; Eldar와 Elowitz, 2010). 반면에 세포의 클론 집단은 측정 된 시간에 세포 당 주어진 단백질 발현 수준에서 이질성을 나타냅니다 (Chang et al., 2008). 확률 성과 이질성은 세포 운명 다양 화, 표현형 변이 및 세포 내 신호 증폭에 필수적입니다 (Locke et al., 2011; Selvarajoo, 2012).
확률 적 변동 또는 내재적 잡음은 원인 분자 종의 발현은 시간과 세포 사이에서 변하여 상관 관계가없는 반응을 유발합니다 (Elowitz et al., 2002). 이것은 카피 수가 적은 mRNA와 단백질에서 특히 두드러집니다. 따라서 고유 노이즈로 인해 샘플 (셀) 간의 상관 관계를 낮출 수 있습니다 (그림 1A). 외인성 요인으로 인한 생물학적 소음의 다른 원인으로는 세포 크기의 가변성, 분자 카피 수 및 개별 세포 간의 환경 변동이 있습니다.이러한 요인은 결정 론적 중심 교리를 왜곡하고 강한 상관 관계를 약한 상관 관계로 바꿀 가능성이 높습니다 (그림 1B).
최근 한 연구는 단일 분자 수준에서 개별 세포 사이의 대장균 mRNA와 단백질 발현을 비교하고 중심 교리에 깊이 질문하는 시나리오를 제공했습니다. Taniguchi et al. (2010)은 단일 세포에서 개별 tufA mRNA와 단백질 수준 사이에 상관 관계 (R2 ~ 0)가 없음을 밝혔다. 특히, 그들은 mRNA와 단백질 수명의 차이로 인해 상관 관계가 없을 가능성이 있다고 결론지었습니다. 이것은 그럴듯한 설명이지만 Taniguchi et al. mRNA 수준의 시간 평균이 단백질 수준과 연관되어야한다고 주장함으로써 장기 보유 가설을 반증하지 않도록주의했습니다. 그러나 이것이 실제 사례임을 입증하는 증거는 없으며 Taniguchi et al.에서 상호 정보를 사용하여 비선형 종속성을 평가했을 때 (Steuer et al., 2002; Tsuchiya et al., 2010). 즉, I ~ 0이라는 결과가 비의존적임을 발견했습니다. 이는 단일 분자 수준에서 개별 세포 간의 단백질 발현에 대한 mRNA가 분명히 무관하다는 것을 확인시켜줍니다. 또한 상관 관계 플롯에서 단일 분자 수준으로 확대하면 쌍별 상관 관계가 약하다는 것이 분명합니다 (그림 1A, 삽입, 그림 참조).
특히 세포 집단 수준에서 Taniguchi et al. R2 = 0.29로 mRNA와 단백질 발현 사이에 상대적으로 높은 상관 관계를 보여줄 수있었습니다 (그림 2A). 실제로 Lu et al. (2007), E. coli 집단에 대해서도 상대적으로 높은 상관 관계를 보였습니다 (R2 = 0.47). Saccharomyces cerevisiae (Futcher et al., 1999), 뮤린 NIH / 3T3 섬유 아세포 (Schwanhäusser et al., 2011) 및 여러 다른 세포 집단 (Nie et al., 2006; Schmidt et al., 2007; Jayapal et al. al., 2008; de Sousa Abreu et al., 2009) 모두 transcriptome-wide와 proteome-wide 표현 사이의 상관 관계를 보여주었습니다 (표 1). 그렇다면 단일 세포에서 개별 mRNA와 단백질 발현 사이에는 상관 관계가 없는데, 집단 수준에서는 대규모 mRNA와 단백질 발현간에 집단적 관계가 관찰되는 이유는 무엇입니까?
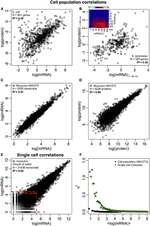
그림 2. 오 믹스 차원의 표현 상관 관계. 세포 집단 : t = 60 분에서의 mRNA 발현과 t에서의 단백질 발현 사이의 (A) E. coli (Taniguchi et al., 2010) 및 (B) S. cerevisiae (Fournier et al., 2010)에서 mRNA- 단백질 상관 관계 = 360 분. 삽입 : 모든 시점 간의 상관 관계 매트릭스는 mRNA와 단백질 간의 상관 관계가 지연된 증가를 보여줍니다. (C) 뮤린 NIH / 3T3 세포의 두 샘플 사이의 mRNA 및 (D) 단백질 발현 (Schwanhäusser et al., 2011). 단일 세포 : (E) 두 난 모세포 사이의 mRNA 발현 (Tang et al., 2009). 빨간색 점선은 낮은 mRNA 발현 영역을 나타냅니다 (log (mRNA) < 5). (F) 세포 집단 (NIH / 3T3, black dots, Schwanhäusser et al., 2011) 및 단일 세포 (Oocytes, green triangles, Tang et al., 2009)에 대한 소음 (η2) 대 log (mRNA 발현). 각 점은 P = 100 mRNA 그룹의 값을 나타냅니다. η2는 모든 mRNA 발현에 대한 세포 집단에 대해 거의 0에 가깝습니다. 단일 세포의 경우 η2는 카피 수가 가장 낮은 mRNA에서 가장 높고 더 높은 카피 수에서는 0에 가까워집니다.
우리는 차이점에는 두 가지 주요 이유가 있습니다. 첫째, 앞서 언급했듯이 자연적으로 생물학적이든 비 생물 적이든 소음은 상관 관계를 감소시킵니다. 단일 세포에 대한 분석은 확률 성과 가변성의 중요성을 보여 주었으므로 이러한 효과는 단일 세포 상관 관계를 줄이는 데 중요합니다. 앙상블 수준에서 세포가 모집단으로 샘플링되면 무작위 노이즈가 모든 범위의 분자 표현 (그림 1C–F)에서 상쇄되므로 전체 (내재 + 외) 노이즈가 감소하여 평균 반응과 자기 조직화가 나타납니다 (그림 1C–F). Karsenti, 2008; Selvarajoo, 2011; Hekstra 및 Leibler, 2012; Selvarajoo 및 Giuliani, 2012). 따라서, 상당한 정도의 mRNA- 단백질 발현 상관 관계가 나타난다. 둘째, 단일 세포 연구 (Taniguchi et al., 2010)의 경우 개별 mRNA- 단백질 발현 상관 관계를 수많은 세포에서 비교했습니다. 그러나 세포 집단 연구에서 비교는 세포 사이의 단일 분자에서 발견되는 발현 범위보다 몇 배 더 큰 수천 개의 mRNA와 단백질에 대해 전체적으로 이루어집니다. 따라서 단일 분자 변이의 영향이 무시할 수있게됨에 따라 집단 수준에서 더 높은 상관 관계가 나타납니다.
세포 집단에 대해 상관 관계가 관찰 되었음에도 불구하고 완벽한 상관 관계에서 큰 편차가 발생하는 확실한 이유가 있습니다.앞서 언급했듯이, 한 가지 요점은 mRNA와 단백질이 여러 가지 누락 된 과정과 함께 순차적으로 위치하며 중앙 교리에서는 나타나지 않는다는 것입니다. 누락 된 중간체를 생화학 적 경로를 따라 추가하면 정보 흐름이 눈에 띄게 지연되고 (Selvarajoo, 2006, 2011; Piras et al., 2011) 결과적으로 이들 간의 상관 관계가 악화 될 수 있습니다. 이것은 또한 Taniguchi 등이 지적한 사실의 일부일 수 있습니다. mRNA와 단백질 발현은 수명이 다릅니다. 특히,이 가정은 mRNA- 단백질 발현의 시간적 상관 관계가 초기에 낮았 음을 보여준 Rapamycin으로 처리 된 S. cerevisiae에 대한 최근 연구에서 뒷받침됩니다. 그럼에도 불구하고 40 분에 R2 = 0.01이지만 섭동 후 360 분에 걸쳐 상관 관계가 증가했습니다. R2 = 0.36 (Fournier et al., 2010, 그림 2B). 데이터는 화학적 섭동시 mRNA와 단백질 발현 사이의 초기 반응이 시간 지연과 그들 사이의 다른 운동 메커니즘뿐만 아니라자가 분비 또는 파라 크린 신호 간섭과 같은 2 차 효과로 인해 편차가 있음을 나타냅니다 (Shvartsman et al., 2002; Isalan et al., 2008). 시간이 지남에 따라 섭동의 영향이 약화되면 상관 관계의 회복이 발생했습니다.
mRNA- 단백질 상관 관계를 감소시키는 데 순차적 지연 프로세스 또는 다른 수명이 중요하다는 가정을 추가로 확인하기 위해 R2를 비교했습니다. 세포 집단 및 단일 세포에서 중심 교리의 동일한 분자 종 (예 : mRNA와 mRNA 사이). NIH / 3T3 복제물 (Schwanhäusser et al., 2011) (그림 2C)과 Mycobacterium tuberculosis (Ward et al., 2008) 세포 집단 샘플 간의 전 사체 전체 mRNA-mRNA 발현 상관 관계는 R2
0.9 (표 1). NIH / 3T3 세포 (Schwanhäusser et al., 2011) (그림 2D), Porphyromonas gingivalis (Xia et al., 2007) 및 Glycine max (Brandão et al. , 2010) (표 1). 동일한 종을 비교하는 이러한 데이터는 매우 높은 상관 관계를 생성하기 때문에 순차적 인 지연 과정이나 다른 수명이 mRNA와 단백질 발현 사이의 집단 수준 상관 관계 구조를 낮추는 원인이 될 수 있습니다.
단일 쥐 난 모세포 ( Tang et al., 2009), 전체 mRNA-mRNA 발현을 비교할 때 고도로 상관 된 구조가 관찰됩니다 (R2 = 0.92, 그림 2E). 그러나 낮은 발현 mRNA (대수식 < 5 사용)에만 초점을 맞추면 확률 적 잡음은 쌍별 상관 관계를 상당히 낮 춥니 다 (R2 < 0.54). 이 결과를 조사하기 위해 전체 mRNA 발현에 걸쳐 잡음 η2 = σ2XY / μ2XY를 평가했습니다 (그림 2F). η2는 표현과 비교하여 확률 적 변동의 뚜렷한 효과로 인해 가장 낮은 표현에서 가장 높고, 이러한 노이즈가 덜 중요 해지는 높은 표현에서는 0에 접근합니다 (Piras et al., 2012). 예상대로 세포 집단의 경우 무작위 노이즈 제거로 인해 전체 발현 범위에서 거의 0에 가까운 노이즈가 관찰됩니다 (그림 1E, F).
전체 mRNA-mRNA 발현에 대해 높은 상관 관계가있는 구조도있었습니다. R2 ~ 0.7에서는 덜 중요하지만 단일 암 세포에 대해보고되었습니다 (Fan et al., 2012) (표 1). 더욱이 LPS로 자극 된 인간 대 식세포의 단백질-단백질 발현 비교는 R2 ~ 0.72로 높은 상관 관계를 보였다 (Shin et al., 2011) (표 1). 단일 세포에서 개별 mRNA- 단백질 발현 간에는 상관 관계가 없지만 단일 세포에서 동일한 분자 종 간의 대규모 또는 오 믹스 차원의 상관 관계는 매우 높습니다.
따라서 단일 세포이든 세포 집단이든 상관 없습니다. omics 전체 데이터는 동일한 분자 종 (mRNA 대 mRNA, 단백질 대 단백질) 간의 상관 관계가 다른 종 (mRNA 대 단백질)보다 현저하게 높다는 것을 나타냅니다. 이것은 시간 지연 과정과 다른 수명이 상관 관계를 줄이는 데 핵심이되지만 이러한 메커니즘은 단백질 발현에 대한 단일 세포 “개별 전 사체간에 관찰되는 상관 관계 구조의 부족을 지원하기에 충분하지 않다는 사실을 반영합니다.
그래서 지금까지 다양한 세포 시스템의 mRNA와 단백질의 대규모 발현을 조사한 결과 상관 관계 구조가 전 세계적으로 나타나는 것으로 나타 났지만, 상관 관계 분석은 두 개의 테스트 된 샘플 사이의 연결성 만 드러내고 그 방향은 보여주지 않았습니다. 중앙 교리가 전 세계적으로 유효하려면 정보의 전체 흐름이 DNA에서 단백질로 이루어져야합니다. 이러한 정보의 흐름은 세포 집단의 수용체를 교란시키고 그 결과를 모니터링하는 것을 포함하는 수많은 다른 연구에서 입증되었습니다. DNA에 결합하는 전사 인자의 역학 및 대규모 유전자 발현 유도 (그림 3A).예를 들어, LPS로 자극 된 면역 세포의 경우, 전사 인자 NF-κB의 활성화가 약 15 분에 발생한다는 것이 입증되었습니다 (Liu et al., 1999), 그 하류 유전자는 약 30 분에 유도됩니다. min (Liu et al., 1999; Xaus et al., 2000; Selvarajoo et al., 2008) 및 60–90 분 영역에서 해당 단백질의 번역 (Kawai et al., 1999; Xaus et al. ., 2000) (그림 3B). 번역 정보 흐름에 대한 전체 전사의 이러한 순차적 인 방향은 세포 집단 수준에서 대장균과 같은 박테리아 시스템에서도 관찰됩니다 (Golding et al., 2005).

그림 3. 중앙 교리의 정보 흐름. (A) 선형 정보 흐름에 따른 전사 인자 NF-κB 및 tnf 유전자를 통한 LPS / TLR4 유도 TNF 발현의 개략도. (B) 세포 집단 수준에서 NF-κB (상단 패널), tnf (중간 패널) 및 TNF (하단 패널) 발현의 프로모터 결합 활성의 실험적 시간 프로파일. (C) 단일 세포 수준에서 프로모터 역학, mRNA 및 단백질 발현의 도식적 시간 프로파일 (Raj 및 van Oudenaarden, 2009).
또는 단일 세포 분해능에 대한 조사는 선형 정보 흐름에 대한 임의의 변동을 나타냅니다. DNA 프로모터 영역에 결합하는 전사 인자는 양자 적이며 결과적으로 mRNA 전사의 파열 동작을 유발하고 결과적으로 단백질 번역의 가변성을 유도합니다. 동일한 세포 사이에서도 (그림 3C) (Raj and van Oudenaarden, 2009; Eldar and Elowitz, 2010; Locke et al., 2011; Hekstra and Leibler, 2012; Selvarajoo, 2012). 결과적으로 특정 시점에서 단일 세포에 대한 개별 분자 반응은 인구 평균 규모에 비해 다소 시끄 럽습니다 (Selvarajoo, 2011).
결론
에 표시된 예 이 논문은 세포 집단과 단일 세포에 대한 중앙 교리의 종간에 관찰 된 상관 관계 값의 순서 차이를 강조합니다. 세포 집단의 통계 분석은 동일한 분자 종 간의 발현 상관 관계가 매우 높고 종 간의 발현 상관 관계가 적당히 높다는 것을 보여줍니다. 동일한 종 간의 단일 세포 상관 관계는 세포 집단과 비슷하지만 생물학적 소음의 뚜렷한 효과로 인해 특히 카피 수가 적은 전 사체의 경우 표현 플롯에서 더 넓은 산포를 나타 냈습니다. 특히, 단일 세포의 쌍별 상관 관계는 개별 분자에 대해 0이됩니다 (Taniguchi et al., 2010). 사실, 분자 표현의 확률 적 변동과 가변성은 세포 운명 결정을 생성하고 세포 상태를 기울이는 데 기능하는 것으로 알려져 있습니다 (Losick and Desplan, 2008; Eldar and Elowitz, 2010; Kuwahara and Schwartz, 2012). 우리는 수천 개의 분자에 걸친 긴밀한 유전자 및 단백질 조절 네트워크의 결과로 강력한 오 믹스 차원의 상관 관계가 발생한다고 믿습니다 (Barabási and Oltvai, 2004; Karsenti , 2008) 결과 평균 반응이 나타납니다. 적은 수 또는 개별 분자를 분석하면 상관 관계 구조를 관찰 할 수 없습니다.
전반적으로 단일 DNA에서 단백질로의 정보 흐름을 보는 것이 핵심 교리에 의문을 제기 할 것이라고 생각할 수 있습니다. 한 번에 각 분자의 반응은 상관 관계가 없을 가능성이 높지만, 전 세계적으로 평균 결정 론적 반응의 관찰은 유전자의 순 평형을 시사합니다. IC 정보는 경로의 맨 오른쪽에 남아 있습니다. 따라서 중심 도그마는 단일 유전자 대 단백질 수준이 아닌 오 믹스 전체 규모의 거시적 세포 정보 흐름으로 간주되어야합니다. 따라서 우리는 그 단순함이 계속해서 살아있는 시스템의 가장 영향력있는 이론적 기둥 중 하나로 남아있을 것이라고 믿습니다.
이해 상충 선언문
저자는 연구가 다음에서 수행되었다고 선언합니다. 잠재적 인 이해 상충으로 해석 될 수있는 상업적 또는 재정적 관계의 부재.
감사합니다
Kentaro Hayashi는 의견에 감사드립니다. 쓰루 오카시와 야마가타 현의 연구비는 그들의 지원에 감사드립니다.
Crick, F. (1958). 단백질 합성에. Symp. Soc. 특급. Biol. 12, 139–163.
Pubmed Abstract | 공개 된 전체 텍스트
Crick, F. (1970). 분자 생물학의 중심 교리. Nature 227, 561–563.
Pubmed Abstract | 공개 된 전체 텍스트
Hayden, E. C. (2011). 변경된 RNA의 증거는 논쟁을 불러 일으 킵니다. Nature 473, 432.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Hekstra, D. R. 및 Leibler, S. (2012). 복제 미생물 폐쇄 생태계의 비상 및 통계법. Cell 149, 1164–1173.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Kuwahara, H. 및 Schwartz, R. (2012).mRNA 분해 제어를 통한 유전자 발현 과정에서 확률 적 정상 상태 획득. J. R. Soc. 인터페이스 9, 1589–1598.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Nie, L., Wu, G. 및 Zhang, W. (2006). Desulfovibrio vulgaris에서 번역 효율과 관련된 여러 서열 특징에 의해 영향을받는 mRNA 발현 및 단백질 풍부도의 상관 관계 : 정량 분석. Genetics 174, 2229–2243.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Prusiner, S. B. (1998). 프리온. Proc. Natl. Acad. Sci. U.S.A. 95, 13363–13383.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Rosner, B. (2011). 생물 통계학의 기초. 7th Edn. 매사추세츠 주 보스턴 : Duxbury Press.
Selvarajoo, K. (2006). MyD88 녹아웃에서 수신자와 유사한 수용체 4 신호 전달 경로의 차동 활성화 기계 발견. FEBS Lett. 580, 1457–1464
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Selvarajoo, K. (2011). 거시적 보존 법칙은 수신자와 유사한 수용체 신호의 인구 역학에서 밝혀졌습니다. Cell Commun. 신호. 9, 9.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Selvarajoo, K. (2012). 단일 세포 및 집단 역학에서 다중 생물학적 결정을 이해합니다. Wiley Interdiscip. 목사 Syst. Biol. Med. 4, 385–399.
Pubmed Abstract | Pubmed 전체 텍스트 | CrossRef 전체 텍스트
Stewart, T. R. (1990). 상관 계수의 분해 및 예측 기술 분석에 사용. 일기 예보. 5, 661 ~ 666.