El procesamiento de la información es fundamental en todos los campos de la ciencia. En biología molecular, el dogma central, acuñado por primera vez por Francis Crick (Crick, 1958, 1970), es una columna vertebral clásica de las células vivas para ejecutar fundamentalmente procesos desde la división celular hasta la muerte a través de las vías de información de ADN, ARN y proteínas. Más específicamente, el dogma central describe la transferencia de información de secuencia durante la replicación del ADN, la transcripción en ARN y la traducción en cadenas de aminoácidos que forman proteínas. Al mismo tiempo, también establece que la información no puede fluir de una proteína a otra o de un ácido nucleico.
Desde el advenimiento de los enfoques sistémicos y de alto rendimiento durante las últimas dos décadas, estos pasos amplios, que no incluyen Los complejos detalles regulatorios han sido objeto de un intenso escrutinio. Las características reguladoras faltantes, como los mecanismos de corrección / reparación del ADN y el empalme alternativo de pre-ARNm, introducen varios pasos intermedios. Estos pasos adicionales interfieren con los pasos clave del dogma y probablemente alteren la dinámica de la información. Además, la epigenética, o el papel que juegan las estructuras de la cromatina, la metilación del ADN y las modificaciones de las histonas, también parecen ir en contra de las vías simples del dogma (Shapiro, 2009; Luco et al., 2011). El empalme de proteínas, o la capacidad de una proteína (inteínas) para alterar su propia secuencia, descubierto en tiempos recientes (Volkmann y Mootz, 2012) y los priones, que modifican otras secuencias de proteínas (Prusiner, 1998), evitan la vía de transferencia de información del dogma. Otras investigaciones informaron errores o desajustes entre las secuencias de ARN y su ADN codificante (Hayden, 2011; Li et al., 2011). Tomados en conjunto, estos datos arrojan dudas sobre la validez del dogma central en el contexto de la ciencia actual y, por lo tanto, cuestionan la simplicidad del flujo de información lineal (ADN a ARN y ARN a proteína).
Para poner las cosas en perspectiva, necesitamos herramientas analíticas que investiguen las inquietudes o discrepancias con respecto a la teoría de larga data. Una técnica simple pero muy útil para buscar propiedades globales en conjuntos de datos de alto rendimiento es el análisis de correlación estadística, que se ha utilizado ampliamente y con éxito para observar patrones en sistemas complejos como el clima (Stewart, 1990), los mercados de valores (Lo y MacKinlay). , 1988) y cosmología (Amati et al., 2008). Hay varios tipos de análisis de correlación que evalúan dependencias lineales (por ejemplo, producto-momento de Pearson) y no lineales (por ejemplo, rango de Spearman, información mutua) (Steuer et al., 2002; Rosner, 2011). , el análisis de correlación producto-momento de Pearson se ha convertido en el más popular debido a su capacidad para mostrar la estructura organizacional en la forma más simple.
En biología, se han realizado numerosos trabajos que han estudiado las correlaciones en el ARNm y datos de expresión de proteínas (ver más abajo y la Tabla 1). En teoría, cuando se comparan dos muestras que contienen datos de alta dimensión (como microarrays y proteómicos), los análisis de correlación proporcionan una medida de desviación de la unidad como fuente de diferencia entre las muestras . Brevemente, dos muestras con información idéntica y completamente no idéntica mostrarán correlación unitaria (R2 = 1) y nula (R2 = 0), respectivamente.
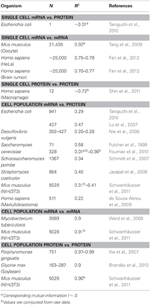
Tabla 1. ARNm y proteína expr ession correlaciones en varios organismos.
La correlación perfecta (R2 = 1) es una situación idealizada que está lejos de la realidad, ya que o el ruido experimental solo interfiere y reduce la correlación. Además, los últimos años han puesto de relieve la existencia de ruido biológico: los estudios sobre células y moléculas individuales han mostrado estocasticidad en la dinámica de expresión génica debido al efecto combinatorio de un bajo número de copias moleculares y la naturaleza cuántica de la dinámica del promotor (Raj y van Oudenaarden, 2009; Eldar y Elowitz, 2010). Por otro lado, las poblaciones clonales de células muestran heterogeneidad en los niveles de expresión de una proteína determinada por célula en cualquier momento medido (Chang et al., 2008). Juntas, la estocasticidad y la heterogeneidad son esenciales para producir la diversificación del destino celular, las variaciones fenotípicas y la amplificación de las señales intracelulares (Locke et al., 2011; Selvarajoo, 2012).
Las fluctuaciones estocásticas, o ruido intrínseco, causan la expresión de una especie molecular varía en el tiempo y entre células, lo que conduce a respuestas no correlacionadas (Elowitz et al., 2002). Esto es especialmente importante para los ARNm y las proteínas con un bajo número de copias. Por lo tanto, la correlación entre muestras (células) se puede reducir debido al ruido intrínseco (Figura 1A). Otras fuentes de ruido biológico debido a factores extrínsecos incluyen la variabilidad en el tamaño de las células, el número de copias moleculares y las fluctuaciones ambientales entre las células individuales.Estos factores distorsionan el dogma central determinista y probablemente alteren correlaciones fuertes en otras más débiles (Figura 1B).
Un estudio reciente comparó el ARNm de Escherichia coli y las expresiones de proteínas entre células individuales a nivel de molécula única y proporcionó un escenario que cuestiona profundamente el dogma central. Taniguchi y col. (2010) revelaron que no existe correlación (R2 ~ 0) entre los niveles de ARNm de tufA individual y de proteínas en células individuales. En particular, llegaron a la conclusión de que la falta de correlación probablemente se deba a diferencias en la vida útil del ARNm y las proteínas. Aunque esta es una explicación plausible, Taniguchi et al. Tuvieron cuidado de no refutar la hipótesis de larga data al afirmar que los promedios temporales de los niveles de ARNm deberían correlacionarse con los niveles de proteínas. Sin embargo, no se mostró evidencia que demuestre que este es el caso real, y cuando evaluamos las dependencias no lineales utilizando información mutua (Steuer et al., 2002; Tsuchiya et al., 2010) en Taniguchi et al. conjunto de datos, encontramos que el resultado no es dependiente, es decir, I ~ 0. Esto confirma que las expresiones de ARNm a proteínas entre células individuales a nivel de molécula única no están claramente relacionadas. Además, al hacer zoom a nivel de una sola molécula en el gráfico de correlación, es evidente que sus correlaciones por pares son débiles (Figura 1A, inserto, para ilustración).
En particular, a nivel de población celular, Taniguchi et al. Alabama. fueron capaces de mostrar una correlación relativamente alta entre el ARNm y las expresiones de proteínas con R2 = 0,29 (Figura 2A). De hecho, otro estudio independiente de Lu et al. (2007), para la población de E. coli, también mostró una correlación relativamente alta (R2 = 0,47). Análisis similares realizados en Saccharomyces cerevisiae (Futcher et al., 1999), fibroblasto NIH / 3T3 murino (Schwanhäusser et al., 2011) y varias otras poblaciones celulares (Nie et al., 2006; Schmidt et al., 2007; Jayapal et al., 2011). al., 2008; de Sousa Abreu et al., 2009) mostraron estructuras correlacionadas entre las expresiones de todo el transcriptoma y de todo el proteoma (Tabla 1). Entonces, ¿por qué no hay correlación entre las expresiones de ARNm y proteínas individuales en células individuales, mientras que a nivel de población, se observan relaciones colectivas entre las expresiones de ARNm y proteínas a gran escala?
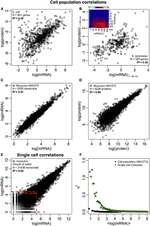
Figura 2. Correlaciones de expresión en todo el ómic. Poblaciones celulares: correlaciones de ARNm-proteína en (A) E. coli (Taniguchi et al., 2010) y (B) S. cerevisiae (Fournier et al., 2010) entre expresiones de ARNm en t = 60 min y expresiones de proteínas en t = 360 min. Insertar: la matriz de correlación entre todos los puntos temporales muestra un aumento retardado de las correlaciones entre el ARNm y las proteínas. (C) ARNm y (D) expresiones de proteínas entre dos muestras de células NIH / 3T3 murinas (Schwanhäusser et al., 2011). Células individuales: (E) Expresiones de ARNm entre dos ovocitos (Tang et al., 2009). Las líneas de puntos rojas indican las regiones de bajas expresiones de ARNm (log (ARNm) < 5). (F) Ruido (η2) versus log (expresiones de ARNm) para la población celular (NIH / 3T3, puntos negros, Schwanhäusser et al., 2011) y células individuales (Ovocitos, triángulos verdes, Tang et al., 2009). Cada punto representa el valor de un grupo de P = 100 mRNA. η2 está cerca de cero para la población celular para todas las expresiones de ARNm. Para células individuales, η2 es más alto para los ARNm con los números de copia más bajos y se acerca a cero para números de copias más altos.
Creemos hay dos razones principales para las diferencias. En primer lugar, como se señaló anteriormente, el ruido, ya sea de naturaleza biológica o no biológica, reduce la correlación. Dado que los análisis de celdas individuales han demostrado la importancia de la estocasticidad y la variabilidad, estos efectos son cruciales para reducir las correlaciones de celdas individuales. A nivel de conjunto, cuando las células se muestrean en una población, el ruido total (intrínseco + extrínseco) se reduce, ya que el ruido aleatorio se cancela en todo el rango de expresiones moleculares (Figuras 1C-F), para revelar la respuesta promedio y la autoorganización ( Karsenti, 2008; Selvarajoo, 2011; Hekstra y Leibler, 2012; Selvarajoo y Giuliani, 2012). Por tanto, surge un buen grado de correlación entre la expresión de ARNm y proteína. En segundo lugar, para el estudio de una sola célula (Taniguchi et al., 2010), se comparó la correlación de expresión de ARNm-proteína individual en numerosas células. Sin embargo, en los estudios de población celular, la comparación se realiza en su totalidad, a través de miles de ARNm y proteínas en varios órdenes de magnitud mayores que el rango de expresión encontrado para una sola molécula entre células. Esto, por lo tanto, conduce a correlaciones más altas a nivel de población ya que el efecto de variaciones moleculares individuales se vuelve insignificante.
A pesar de que se observan estructuras correlacionadas para poblaciones celulares, existen razones tangibles para la gran desviación de la correlación perfecta.Como se señaló anteriormente, un punto clave es que los ARNm y las proteínas se ubican secuencialmente con varios procesos faltantes, no representados en el dogma central. Agregar los intermedios faltantes a lo largo de una ruta bioquímica provocará un retraso notable en el flujo de información (Selvarajoo, 2006, 2011; Piras et al., 2011), y la correlación entre ellos podría verse afectada como resultado. Esto también podría ser parte del hecho señalado por Taniguchi et al. que las expresiones de ARNm y proteínas tienen vidas diferentes. En particular, esta postulación está respaldada en un trabajo reciente sobre S. cerevisiae tratada con rapamicina que mostró que las correlaciones temporales de la expresión de la proteína-ARNm eran inicialmente bajas, R2 = 0.01 a los 40 min, sin embargo, más de 360 min después de la perturbación, la correlación aumentó, R2 = 0.36 (Fournier et al., 2010, Figura 2B). Los datos indican que ante la perturbación química, la respuesta inicial entre el ARNm y las expresiones de proteínas se desvía debido al retardo del tiempo y a los diferentes mecanismos cinéticos entre ellos, así como a efectos secundarios como la interferencia de señalización autocrina o paracrina (Shvartsman et al., 2002; Isalan et al., 2008). Cuando los efectos de la perturbación se atenúan con el tiempo, se produce la recuperación de las correlaciones.
Para verificar más a fondo la postulación de que los procesos de retardo secuencial o diferentes tiempos de vida son cruciales para disminuir las correlaciones de ARNm-proteína, comparamos R2 entre los mismas especies moleculares del dogma central (por ejemplo, entre ARNm y ARNm) en poblaciones celulares y células individuales. La correlación de expresión de ARNm-ARNm en todo el transcriptoma entre las réplicas de NIH / 3T3 (Schwanhäusser et al., 2011) (Figura 2C) y las muestras de población celular de Mycobacterium tuberculosis (Ward et al., 2008) son muy altas, con R2 > 0.9 (Tabla 1). Estas fuertes correlaciones también se observan entre muestras de población para expresiones proteína-proteína en células NIH / 3T3 (Schwanhäusser et al., 2011) (Figura 2D), Porphyromonas gingivalis (Xia et al., 2007) y Glycine max (Brandão et al. , 2010) (Tabla 1). Dado que estos datos que comparan las mismas especies producen correlaciones muy altas, es concebible que los procesos de retardo secuencial o las diferentes vidas sean responsables de reducir las estructuras de correlación a nivel de población entre las expresiones de ARNm y proteínas.
En ovocitos murinos individuales ( Tang et al., 2009), al comparar expresiones completas de ARNm-ARNm, se observa una estructura altamente correlacionada (R2 = 0,92, Figura 2E). Sin embargo, al centrarse solo en los ARNm de baja expresión (con expresiones logarítmicas < 5), el ruido estocástico reduce la correlación por pares de manera bastante drástica (R2 < 0,54). Para probar este resultado, evaluamos el ruido, η2 = σ2XY / μ2XY, a través de expresiones de ARNm completas (Figura 2F). Observamos que η2 es más alto para las expresiones más bajas, debido al efecto pronunciado de las fluctuaciones estocásticas en comparación con sus expresiones, y se acerca a cero para las expresiones más altas, donde dicho ruido se vuelve menos significativo (Piras et al., 2012). Para la población celular, como se esperaba, se observa ruido cercano a cero en todo el rango de expresión debido a la cancelación del ruido aleatorio (Figuras 1E, F).
También se observaron estructuras altamente correlacionadas para las expresiones de ARNm-ARNm completo reportados para una sola célula cancerosa (Fan et al., 2012), aunque menos significativo con R2 ~ 0.7 (Tabla 1). Además, la comparación de expresiones de proteína-proteína en macrófagos humanos estimulados con LPS también mostró correlaciones altas, R2 ~ 0,72 (Shin et al., 2011) (Tabla 1). Aunque no existe una correlación entre las expresiones de la proteína de ARNm individuales en células individuales, la correlación a gran escala u omics amplia entre las mismas especies moleculares en células individuales es muy alta.
Por lo tanto, ya sean células individuales o poblaciones de células , los datos de todo el ómico indican que las correlaciones entre la misma especie molecular (ARNm frente a ARNm y proteína frente a proteína) son notablemente más altas que entre diferentes especies (ARNm frente a proteína). Esto refleja el hecho de que, aunque los procesos de retardo de tiempo y las diferentes vidas útiles son clave para reducir las correlaciones, estos mecanismos no son suficientes para respaldar la falta de estructura de correlación observada entre la transcripción individual de células individuales a las expresiones de proteínas.
Entonces Hasta ahora, a través de la investigación de expresiones a gran escala de ARNm y proteínas de varios sistemas celulares, hemos demostrado que las estructuras de correlación emergen a escala global. Sin embargo, los análisis de correlación revelan solo la conectividad entre dos muestras analizadas y no muestran la dirección de flujo de información. Para que el dogma central sea válido a escala global, el flujo general de información debe ser del ADN a las proteínas. Este flujo de información ha sido demostrado por una miríada de otros estudios que involucran la perturbación de los receptores de las poblaciones celulares y el monitoreo de la información resultante. dinámica de los factores de transcripción que se unen al ADN y la inducción de expresiones génicas a gran escala (Figura 3A).Por ejemplo, en el caso de las células inmunes estimuladas con LPS, se ha demostrado que la activación del factor de transcripción NF-κB se produce alrededor de los 15 minutos (Liu et al., 1999), la inducción de sus genes aguas abajo alrededor min (Liu et al., 1999; Xaus et al., 2000; Selvarajoo et al., 2008), y la traducción de las proteínas correspondientes en la región de 60-90 min (Kawai et al., 1999; Xaus et al. ., 2000) (Figura 3B). Esta dirección secuencial de la transcripción general al flujo de información de traducción también se observa para sistemas bacterianos, como E. coli, a nivel de población celular (Golding et al., 2005).

Figura 3. El flujo de información del dogma central. (A) Esquema de la expresión de TNF inducida por LPS / TLR4, a través del factor de transcripción NF-κB y el gen tnf, siguiendo el flujo de información lineal. (B) Perfiles temporales experimentales de la actividad de unión del promotor de expresiones de NF-κB (paneles superiores), tnf (paneles intermedios) y TNF (paneles inferiores) a nivel de población celular. (C) Perfiles temporales esquemáticos de la dinámica del promotor, el ARNm y las expresiones de proteínas a nivel de una sola célula (Raj y van Oudenaarden, 2009).
Alternativamente, las investigaciones a la resolución de una sola célula revelan fluctuaciones aleatorias sobre el flujo de información lineal: los factores de transcripción que se unen a las regiones promotoras del ADN son cuánticos, lo que da como resultado un comportamiento explosivo de la transcripción del ARNm y, posteriormente, induce la variabilidad en la traducción de la proteína. incluso entre células idénticas (Figura 3C) (Raj y van Oudenaarden, 2009; Eldar y Elowitz, 2010; Locke et al., 2011; Hekstra y Leibler, 2012; Selvarajoo, 2012). Como resultado, en cualquier momento en particular, la respuesta molecular individual para células individuales es bastante ruidosa en comparación con la escala promedio de la población (Selvarajoo, 2011).
Conclusiones
Los ejemplos que se muestran en Este artículo destaca las diferencias en el orden de los valores de correlación observados entre especies en el dogma central sobre poblaciones celulares y células individuales. Los análisis estadísticos de poblaciones de células muestran que la correlación de expresión entre la misma especie molecular es muy alta y entre especies es moderadamente alta. Aunque las correlaciones de una sola célula entre la misma especie son comparables con las poblaciones de células, mostraron una dispersión más amplia en sus gráficos de expresión debido al efecto pronunciado del ruido biológico, especialmente para las transcripciones con números de copia bajos. En particular, la correlación por pares de células individuales se vuelve cero para moléculas individuales (Taniguchi et al., 2010). De hecho, se sabe que las fluctuaciones estocásticas y la variabilidad en las expresiones moleculares son funcionales en la generación de decisiones sobre el destino de las células y en los estados celulares de inclinación (Losick y Desplan, 2008; Eldar y Elowitz, 2010; Kuwahara y Schwartz, 2012). Creemos que las fuertes correlaciones ómicas se producen como resultado de estrechas redes reguladoras de genes y proteínas en miles de moléculas (Barabási y Oltvai, 2004; Karsenti , 2008) resultando en respuestas promedio emergentes. Al analizar un número pequeño o moléculas individuales, no se puede observar la estructura de correlación.
En general, es concebible que ver el flujo de información de un solo ADN a una proteína cuestione el dogma central ya que la respuesta de cada molécula en un momento único probablemente no se correlacionará. Sin embargo, globalmente, la observación de la respuesta determinista promedio sugiere que el equilibrio neto de la gineta La información ic permanece en el extremo derecho de las vías. Por lo tanto, el dogma central debe verse como un flujo de información celular macroscópica en una escala ómica amplia, y no en un solo gen a nivel de proteína. Como tal, creemos que su simplicidad seguirá siendo uno de los pilares teóricos más influyentes de los sistemas vivos.
Declaración de conflicto de intereses
Los autores declaran que la investigación se realizó en la ausencia de cualquier relación comercial o financiera que pueda interpretarse como un posible conflicto de intereses.
Agradecimientos
Se agradece a Kentaro Hayashi por sus comentarios. Se agradece su apoyo al fondo de investigación de la ciudad de Tsuruoka y la prefectura de Yamagata.
Crick, F. (1958). Sobre la síntesis de proteínas. Symp. Soc. Exp. Biol. 12, 139-163.
Pubmed Abstract | Texto completo de Pubmed
Crick, F. (1970). Dogma central de la biología molecular. Nature 227, 561–563.
Pubmed Abstract | Texto completo de Pubmed
Hayden, E. C. (2011). La evidencia de ARN alterado suscita debate. Nature 473, 432.
Pubmed Abstract | Texto completo de Pubmed | Texto completo CrossRef
Hekstra, D. R. y Leibler, S. (2012). Leyes de contingencia y estadísticas en ecosistemas microbianos cerrados replicados. Cell 149, 1164-1173.
Pubmed Abstract | Texto completo de Pubmed | Texto completo de CrossRef
Kuwahara, H. y Schwartz, R. (2012).Ganancia estocástica en estado estable en un proceso de expresión génica con control de degradación del ARNm. J. R. Soc. Interface 9, 1589–1598.
Pubmed Abstract | Texto completo de Pubmed | CrossRef Full Text
Nie, L., Wu, G. y Zhang, W. (2006). Correlación de la expresión del ARNm y la abundancia de proteínas afectadas por múltiples características de secuencia relacionadas con la eficiencia de traducción en Desulfovibrio vulgaris: un análisis cuantitativo. Genetics 174, 2229–2243.
Pubmed Abstract | Texto completo de Pubmed | Texto completo CrossRef
Prusiner, S. B. (1998). Priones. Proc. Natl. Acad. Sci. U.S.A. 95, 13363–13383.
Pubmed Abstract | Texto completo de Pubmed | Texto completo CrossRef
Rosner, B. (2011). Fundamentos de bioestadística. 7ª Ed. Boston, MA: Duxbury Press.
Selvarajoo, K. (2006). Descubrimiento de la maquinaria de activación diferencial de las vías de señalización del receptor 4 tipo toll en los knockouts de MyD88. FEBS Lett. 580, 1457–1464.
Pubmed Abstract | Texto completo de Pubmed | Texto completo CrossRef
Selvarajoo, K. (2011). Ley macroscópica de conservación revelada en la dinámica poblacional de la señalización del receptor de peaje. Cell Commun. Señal. 9, 9.
Resumen de Pubmed | Texto completo de Pubmed | Texto completo CrossRef
Selvarajoo, K. (2012). Comprender las decisiones biológicas multimodales de la dinámica de una sola célula y de una población. Wiley Interdiscip. Rev. Syst. Biol. Medicina. 4, 385–399.
Pubmed Abstract | Texto completo de Pubmed | Texto completo CrossRef
Stewart, T. R. (1990). Una descomposición del coeficiente de correlación y su uso para analizar la habilidad de pronosticar. Pronóstico del tiempo. 5, 661–666.