情報処理は科学のすべての分野で不可欠です。分子生物学では、フランシス・クリック(Crick、1958、1970)によって最初に造られた中心的な教義は、DNA、RNA、およびタンパク質情報経路を介して細胞分裂から死までのプロセスを基本的に実行する生細胞の古典的なバックボーンです。より具体的には、セントラルドグマは、DNA複製、RNAへの転写、およびタンパク質を形成するアミノ酸鎖への翻訳中の配列情報の転送を説明します。同時に、情報がタンパク質からタンパク質または核酸に流れることはできないとも述べています。
過去20年間の全身的でハイスループットなアプローチの出現以来、これらの広範なステップには、複雑な規制の詳細は、厳しく監視されています。 DNA校正/修復メカニズムやプレmRNAの選択的スプライシングなどの欠落している規制機能は、いくつかの中間ステップを導入します。これらの追加のステップは、教義の主要なステップを妨害し、情報のダイナミクスを変える可能性があります。さらに、エピジェネティクス、またはクロマチン構造、DNAメチル化、ヒストン修飾が果たす役割も、教義の単純な経路に反しているようです(Shapiro、2009; Luco et al。、2011)。最近発見されたタンパク質スプライシング、またはタンパク質(インテイン)がそれ自体の配列を変更する能力(Volkmann and Mootz、2012)および他のタンパク質配列を修飾するプリオン(Prusiner、1998)は、ドグマ。他の調査では、RNA配列とそれらのコードDNAの間のエラーまたはミスマッチが報告されました(Hayden、2011; Li et al。、2011)。まとめると、これらのデータは、現在の科学の文脈におけるセントラルドグマの有効性に疑問を投げかけ、したがって、線形情報フロー(DNAからRNA、およびRNAからタンパク質)の単純さに疑問を投げかけます。
物事を展望するために、私たちは長年の理論に関する懸念や矛盾を調査する分析ツールを必要としています。ハイスループットデータセット内のグローバルプロパティを検索するためのシンプルでありながら非常に便利な手法の1つは、統計相関分析です。これは、天気(Stewart、1990)、株式市場(LoおよびMacKinlay)などの複雑なシステムのパターンを観察するために広く成功裏に使用されています。 、1988)および宇宙論(Amati et al。、2008)。線形(ピアソンの積率など)と非線形(スピアマンの順位、相互情報量など)の両方の依存関係を評価する相関分析にはいくつかの種類があります(Steuer et al。、2002; Rosner、2011)。 、ピアソンの積率相関分析は、組織構造を最も単純な形式で表示できるため、最も人気があります。
生物学では、mRNAとの相関を研究した多くの研究があります。タンパク質発現データ(以下および表1を参照)。理論的には、高次元(マイクロアレイおよびプロテオミクスなど)データを含む2つのサンプルを比較すると、相関分析により、サンプル間の差異の原因として1からの偏差の測定値が提供されます。簡単に言うと、情報が同一で完全に同一ではない2つのサンプルは、それぞれユニット(R2 = 1)とヌル(R2 = 0)の相関関係を示します。
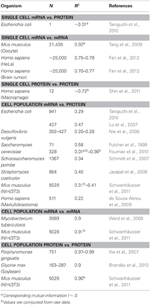
表1.mRNAとタンパク質のexprさまざまな生物におけるession相関。
完全相関(R2 = 1)は、技術的には現実からかけ離れた理想的な状況です。または実験的なノイズだけが干渉し、相関を減らします。さらに、近年、生物学的ノイズの存在が浮き彫りになりました。個々の細胞と分子に関する研究では、低分子コピー数の組み合わせ効果とプロモーターダイナミクスの量子的性質により、遺伝子発現ダイナミクスに確率論が示されています(Raj and van Oudenaarden、 2009; Eldar and Elowitz、2010)。一方、細胞のクローン集団は、測定された任意の時間で、細胞あたりの特定のタンパク質発現のレベルに不均一性を示します(Chang et al。、2008)。一緒に、確率論的および不均一性は、細胞運命の多様化、表現型の変化、および細胞内シグナルの増幅を生み出すために不可欠です(Locke et al。、2011; Selvarajoo、2012)。
確率論的変動、または固有のノイズが原因分子種の発現は時間や細胞間で変化し、無相関の反応を引き起こします(Elowitz et al。、2002)。これは、コピー数が少ないmRNAやタンパク質で特に顕著です。したがって、サンプル(セル)間の相関は、固有のノイズのために低下する可能性があります(図1A)。外因性要因による生物学的ノイズの他の原因には、細胞サイズの変動、分子コピー数、および個々の細胞間の環境変動が含まれます。これらの要因は、決定論的なセントラルドグマを歪め、強い相関関係を弱い相関関係に変える可能性があります(図1B)。
最近のある研究では、大腸菌のmRNAとタンパク質の発現を単一分子レベルで個々の細胞間で比較し、セントラルドグマに深く疑問を投げかけるシナリオを提供しました。谷口ほか(2010)個々のtufA mRNAと単一細胞のタンパク質レベルの間に相関関係(R2〜0)がないことを明らかにしました。特に、彼らは、相関関係の欠如は、mRNAとタンパク質の寿命の違いによる可能性が高いと結論付けました。これはもっともらしい説明ですが、谷口ら。 mRNAレベルの時間平均はタンパク質レベルと相関するはずであると主張することにより、長年の仮説を反証しないように注意しました。しかし、これが実際のケースであることを示す証拠は示されておらず、谷口らの相互情報量を使用して非線形依存性を評価した場合(Steuer et al。、2002; Tsuchiya et al。、2010)。データセットでは、結果が非依存、つまりI〜0であることがわかりました。これにより、単一分子レベルでの個々の細胞間のmRNAからタンパク質への発現が明らかに無関係であることが確認されます。さらに、相関プロットで単一分子レベルでズームすると、それらのペアワイズ相関が弱いことが明らかです(図1A、挿入、図解)。
特に、細胞集団レベルでは、谷口らal。 R2 = 0.29でmRNAとタンパク質の発現の間に比較的高い相関関係を示すことができました(図2A)。実際、Luらによる別の独立した研究。 (2007)、大腸菌集団についても、比較的高い相関を示しました(R2 = 0.47)。 Saccharomyces cerevisiae(Futcher et al。、1999)、マウスNIH / 3T3線維芽細胞(Schwanhäusseretal。、2011)および他のいくつかの細胞集団(Nie et al。、2006; Schmidt et al。、2007; Jayapal etal。 al。、2008; de Sousa Abreu et al。、2009)はすべて、トランスクリプトーム全体とプロテオーム全体の発現の間に相関構造を示しました(表1)。では、なぜ単一細胞での個々のmRNAとタンパク質の発現の間に相関関係がないのに、集団レベルでは、大規模なmRNAとタンパク質の発現の間に集合的な関係が観察されるのでしょうか?
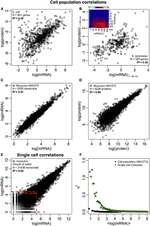
図2.オミック全体の式の相関。細胞集団:(A)E。coli(Taniguchi et al。、2010)および(B)S。cerevisiae(Fournier et al。、2010)におけるt = 60分でのmRNA発現とtでのタンパク質発現の間のmRNA-タンパク質相関= 360分挿入:すべての時点間の相関行列は、mRNAとタンパク質間の相関の遅延した増加を示しています。 (C)マウスNIH / 3T3細胞の2つのサンプル間のmRNAおよび(D)タンパク質の発現(Schwanhäusseretal。、2011)。単一細胞:(E)2つの卵母細胞間のmRNA発現(Tang et al。、2009)。赤い点線は、mRNAの発現が低い領域を示しています(log(mRNA)< 5)。 (F)細胞集団(NIH / 3T3、黒い点、Schwanhäusseretal。、2011)および単一細胞(卵母細胞、緑色の三角形、Tang et al。、2009)のノイズ(η2)対log(mRNA発現)。各ドットは、P = 100mRNAのグループの値を表します。 η2は、すべてのmRNA発現の細胞集団でほぼゼロです。単一細胞の場合、η2はコピー数が最も少ないmRNAで最も高く、コピー数が多い場合はゼロに近づきます。
違いには2つの主な理由があります。第一に、前述のように、生物学的であろうと非生物学的であろうと、ノイズは相関を低下させます。単一セルの分析では確率論と変動性の重要性が示されているため、これらの効果は単一セルの相関を減らすために重要です。アンサンブルレベルでは、細胞が集団にサンプリングされると、ランダムノイズが分子発現のすべての範囲で相殺されるため(図1C〜F)、合計(内因性+外因性)ノイズが減少し、平均応答と自己組織化が明らかになります(図1C〜F)。 Karsenti、2008; Selvarajoo、2011; Hekstra and Leibler、2012; Selvarajoo and Giuliani、2012)。したがって、mRNAとタンパク質の発現にはかなりの相関関係があります。第二に、単一細胞研究(Taniguchi et al。、2010)では、個々のmRNA-タンパク質発現相関が多数の細胞間で比較されました。しかし、細胞集団の研究では、細胞間の単一分子で見られる発現範囲よりも数桁大きい数千のmRNAおよびタンパク質にわたって、全体として比較が行われます。したがって、これにより、単一分子の変動の影響が無視できるようになるため、集団レベルでの相関が高くなります。
細胞集団で相関構造が観察されているにもかかわらず、完全な相関から大きく逸脱する明確な理由があります。先に述べたように、1つの重要なポイントは、mRNAとタンパク質が、セントラルドグマでは表されていない、いくつかの欠落したプロセスとともに順番に配置されていることです。生化学的経路に沿って不足している中間体を追加すると、情報の流れに顕著な遅延が発生し(Selvarajoo、2006、2011; Piras et al。、2011)、結果としてそれらの間の相関が損なわれる可能性があります。これはまた、谷口らによって指摘された事実の一部である可能性があります。そのmRNAとタンパク質の発現は異なる寿命を持っています。特に、この仮定は、ラパマイシンで処理されたS. cerevisiaeに関する最近の研究で支持されており、mRNA-タンパク質発現の時間的相関は最初は低く、40分でR2 = 0.01でしたが、摂動後360分以上で相関が増加しました。 R2 = 0.36(Fournier et al。、2010、図2B)。データは、化学的摂動の際に、mRNAとタンパク質の発現間の初期応答が、時間遅延とそれらの間の異なる速度論的メカニズム、ならびにオートクリンまたはパラクリンシグナル伝達干渉などの二次的影響のために逸脱することを示しています(Shvartsman et al。、2002; Isalan et al。、2008)。摂動の影響が時間の経過とともに減衰すると、相関の回復が起こりました。
mRNA-タンパク質の相関を減少させるには、連続的な遅延プロセスまたは異なる寿命が重要であるという仮定をさらに確認するために、R2を比較しました。細胞集団および単一細胞におけるセントラルドグマの同じ分子種(例えば、mRNAとmRNAの間)。 NIH / 3T3の複製(Schwanhäusseretal。、2011)(図2C)と結核菌(Ward et al。、2008)の細胞集団サンプル間のトランスクリプトーム全体のmRNA-mRNA発現相関は両方とも非常に高く、R2 > 0.9(表1)。このような強い相関関係は、NIH / 3T3細胞(Schwanhäusseretal。、2011)(図2D)、Porphyromonas gingivalis(Xia et al。、2007)およびGlycine max(Brandãoetal。 、2010)(表1)。同じ種を比較するこれらのデータは非常に高い相関関係をもたらすため、連続的な遅延プロセスまたは異なる寿命が、mRNAとタンパク質の発現の間の集団レベルの相関構造を低下させる原因であると考えられます。
単一のマウス卵細胞( Tang et al。、2009)、mRNA-mRNA発現全体を比較すると、相関性の高い構造が観察されます(R2 = 0.92、図2E)。ただし、低発現のmRNA(対数式< 5)のみに焦点を当てると、確率的ノイズはペアワイズ相関を非常に劇的に低下させます(R2 < 0.54)。この結果を精査するために、mRNA発現全体にわたってノイズη2=σ2XY/μ2XYを評価しました(図2F)。 η2は、それらの式と比較して確率的変動の顕著な影響のために、最も低い式で最も高く、そのようなノイズがそれほど重要ではなくなるより高い式ではゼロに近づくことに注意しました(Piras et al。、2012)。細胞集団では、予想通り、ランダムノイズがキャンセルされるため、発現範囲全体でほぼゼロのノイズが観察されます(図1E、F)。
mRNA-mRNA発現全体の相関性の高い構造も単一の癌細胞について報告されていますが(Fan et al。、2012)、R2〜0.7ではそれほど重要ではありません(表1)。さらに、LPSで刺激されたヒトマクロファージにおけるタンパク質間発現の比較でも、R2〜0.72という高い相関関係が示されました(Shin et al。、2011)(表1)。単一細胞における個々のmRNA-タンパク質発現間に相関関係はありませんが、単一細胞内の同じ分子種間の大規模またはオミクス全体の相関関係は非常に高いです。
したがって、単一細胞または細胞集団のどちらでも、オミクス全体のデータは、同じ分子種(mRNA対mRNA、およびタンパク質対タンパク質)間の相関が、異なる種間(mRNA対タンパク質)よりも著しく高いことを示しています。これは、時間遅延プロセスと異なる寿命が相関を減らすための鍵であるが、これらのメカニズムは、タンパク質発現に対する単一細胞の個々の転写物間で観察される相関構造の欠如をサポートするのに十分ではないという事実を反映しています。
これまで、さまざまな細胞系のmRNAやタンパク質の大規模な発現を調査することで、相関構造が地球規模で出現することを示しましたが、相関分析では、2つのテストされたサンプル間の接続性のみが明らかになり、情報の流れ中央のドグマが地球規模で有効であるためには、情報の全体的な流れはDNAからタンパク質まででなければなりません。このような情報の流れは、細胞集団の受容体を混乱させ、結果を監視することを含む他の無数の研究によって実証されています。 DNAに結合する転写因子のダイナミクスと大規模な遺伝子発現の誘導(図3A)。たとえば、LPSで刺激された免疫細胞の場合、転写因子NF-κBの活性化は約15分で起こり(Liu et al。、1999)、その下流遺伝子の誘導は約30分で起こることが実証されています。 min(Liu et al。、1999; Xaus et al。、2000; Selvarajoo et al。、2008)、および60〜90分の領域での対応するタンパク質の翻訳(Kawai et al。、1999; Xaus et al。 。、2000)(図3B)。翻訳情報フローへの全体的な転写のそのような連続的な方向は、細胞集団レベルで、大腸菌などの細菌系でも観察されます(Golding et al。、2005)。

図3.セントラルドグマの情報の流れ。 (A)線形情報フローに従った、転写因子NF-κBおよびtnf遺伝子を介したLPS / TLR4誘導TNF発現の概略図。 (B)細胞集団レベルでのNF-κB(上のパネル)、tnf(中央のパネル)、およびTNF(下のパネル)発現のプロモーター結合活性の実験的時間プロファイル。 (C)単一細胞レベルでのプロモーターダイナミクス、mRNA、およびタンパク質発現の概略的な時間的プロファイル(Raj and van Oudenaarden、2009)。
あるいは、単一細胞の解像度での調査により、線形情報フローのランダムな変動が明らかになります。DNAプロモーター領域に結合する転写因子は量子的であり、mRNA転写のバースト挙動を引き起こし、その後、タンパク質翻訳の変動を誘発します。同一のセル間でも(図3C)(Raj and van Oudenaarden、2009; Eldar and Elowitz、2010; Locke et al。、2011; Hekstra and Leibler、2012; Selvarajoo、2012)。その結果、特定の時点で、単一細胞の個々の分子応答は、母集団の平均スケールと比較してかなりノイズが多くなります(Selvarajoo、2011)。
結論
に示す例この論文は、細胞集団と単一細胞にわたるセントラルドグマの種間で観察された相関値の順序の違いを強調しています。細胞集団からの統計分析は、同じ分子種間の発現相関が非常に高く、種間の発現相関が適度に高いことを示しています。同じ種間の単一細胞の相関関係は細胞集団に匹敵しますが、特にコピー数の少ない転写産物の場合、生物学的ノイズの顕著な影響により、発現プロットでより広いばらつきが見られました。特に、単一細胞のペアワイズ相関は、個々の分子でゼロになります(Taniguchi et al。、2010)。実際、分子発現の確率的変動と変動性は、細胞の運命決定を生成し、細胞状態を傾けるのに機能することが知られています(Losick and Desplan、2008; Eldar and Elowitz、2010; Kuwahara and Schwartz、2012)。数千の分子にわたる緊密な遺伝子およびタンパク質調節ネットワークの結果として、オミクス全体の強い相関関係が生じると考えています(BarabásiおよびOltvai、2004; Karsenti 、2008)結果として平均応答が出現します。少数または個々の分子を分析すると、相関構造を観察できません。
全体として、単一DNAからタンパク質への情報フローを表示すると、中心的なドグマが疑問視されると考えられます。なぜなら、各分子の応答はいつでも相関する可能性が低いからです。しかし、世界的には、平均的な決定論的応答の観察は、遺伝子の正味の平衡がic情報は経路の右端に残ります。したがって、セントラルドグマは、単一の遺伝子からタンパク質レベルではなく、オミクス全体のスケールでの巨視的な細胞情報の流れと見なされるべきです。そのため、そのシンプルさは、生命システムの最も影響力のある理論上の柱の1つとして残り続けると信じています。
利益相反に関する声明
著者は、研究が潜在的な利益相反と解釈される可能性のある商業的または金銭的関係がないこと。
謝辞
林健太郎はコメントに感謝します。鶴岡市と山形県の研究基金の支援に感謝します。
Crick、F。(1958)タンパク質合成について。症状。 Soc。 Exp。 Biol。 12、139–163。
Pubmed Abstract | Pubmed Full Text
Crick、F。(1970)。分子生物学のセントラルドグマ。 Nature 227、561–563。
Pubmed Abstract | Pubmed Full Text
Hayden、E。C.(2011)。変更されたRNAの証拠は議論をかき立てます。 Nature 473、432。
Pubmed Abstract | Pubmed全文| CrossRef全文
Hekstra、D。R.、およびLeibler、S。(2012)。複製微生物閉鎖生態系における不測の事態と統計法則。 Cell 149、1164–1173。
Pubmed Abstract | Pubmed全文| CrossRef全文
桑原秀樹、シュワルツR.(2012)mRNA分解制御を伴う遺伝子発現プロセスにおける確率的定常状態の増加。 J. R.Soc。インターフェイス9、1589〜1598。
Pubmed Abstract | Pubmed全文| CrossRef全文
Nie、L.、Wu、G。、およびZhang、W。(2006)。 Desulfovibrio vulgarisの翻訳効率に関連する複数の配列の特徴によって影響を受けるmRNA発現とタンパク質存在量の相関:定量分析。 Genetics 174、2229–2243。
Pubmed Abstract | Pubmed全文| CrossRef全文
Prusiner、S。B.(1998)プリオン。手順国立Acad。科学U.S.A. 95、13363〜13383。
Pubmed Abstract | Pubmed全文| CrossRef全文
Rosner、B。(2011)。生物統計学の基礎。第7版マサチューセッツ州ボストン:DuxburyPress。
Selvarajoo、K。(2006) MyD88ノックアウトにおけるトール様受容体4シグナル伝達経路の異なる活性化機構の発見。フェブスレター若手580、1457–1464。
Pubmed Abstract | Pubmed全文| CrossRef全文
Selvarajoo、K。(2011) Toll様受容体シグナル伝達の個体群動態で明らかになった巨視的な保存則。セル通信。信号。 9、9。
Pubmed Abstract | Pubmed全文| CrossRef全文
Selvarajoo、K。(2012)単一細胞と個体群動態からのマルチモーダル生物学的決定の理解。ワイリー学際。牧師システム。 Biol。 Med。 4、385–399。
Pubmed Abstract | Pubmed全文| CrossRef全文
Stewart、T。R.(1990)。相関係数の分解と予測スキルの分析におけるその使用。天気予報。 5、661–666。