Le traitement de linformation est essentiel dans tous les domaines de la science. En biologie moléculaire, le dogme central, inventé pour la première fois par Francis Crick (Crick, 1958, 1970), est une épine dorsale classique des cellules vivantes pour exécuter fondamentalement des processus allant de la division cellulaire à la mort en passant par les voies dinformation de lADN, de lARN et des protéines. Plus spécifiquement, le dogme central décrit le transfert dinformations de séquence pendant la réplication de lADN, la transcription en ARN et la traduction en chaînes dacides aminés formant des protéines. Dans le même temps, il indique également que linformation ne peut pas circuler dune protéine à lautre ou à lacide nucléique.
Depuis lavènement des approches systémiques et à haut débit au cours des deux dernières décennies, ces grandes étapes, qui nincluent pas des détails réglementaires complexes ont fait l’objet d’un examen minutieux. Les caractéristiques réglementaires manquantes, telles que les mécanismes de relecture / réparation de lADN et lépissage alternatif du pré-ARNm, introduisent plusieurs étapes intermédiaires. Ces étapes supplémentaires interfèrent avec les étapes clés du dogme et modifient probablement la dynamique de linformation. En outre, lépigénétique, ou le rôle joué par les structures de la chromatine, la méthylation de lADN et les modifications dhistones, semblent également aller à lencontre des voies simples du dogme (Shapiro, 2009; Luco et al., 2011). Lépissage de protéines, ou la capacité dune protéine (intéines) à modifier sa propre séquence, découvert ces derniers temps (Volkmann et Mootz, 2012) et les prions, qui modifient dautres séquences protéiques (Prusiner, 1998), contournent la voie de transfert dinformation du dogme. Dautres enquêtes ont signalé des erreurs ou des inadéquations entre les séquences dARN et leur ADN codant (Hayden, 2011; Li et al., 2011). Prises ensemble, ces données jettent des doutes sur la validité du dogme central dans le contexte de la science actuelle et, par conséquent, remettent en question la simplicité du flux dinformation linéaire (ADN vers ARN et ARN vers protéine).
Pour mettre les choses en perspective, nous avons besoin doutils analytiques qui enquêtent sur les préoccupations ou les divergences concernant la théorie de longue date. Une technique simple mais très utile pour rechercher des propriétés globales dans des ensembles de données à haut débit est lanalyse de corrélation statistique, qui a été largement et avec succès utilisée pour observer des modèles dans des systèmes complexes tels que la météo (Stewart, 1990), les marchés boursiers (Lo et MacKinlay , 1988) et la cosmologie (Amati et al., 2008). Il existe plusieurs types danalyses de corrélation qui évaluent à la fois les dépendances linéaires (par exemple, le moment-produit de Pearson) et non linéaires (par exemple, le rang de Spearman, les informations mutuelles) (Steuer et al., 2002; Rosner, 2011). En particulier , lanalyse de corrélation produit-moment de Pearson est devenue la plus populaire en raison de sa capacité à montrer la structure organisationnelle sous la forme la plus simple.
En biologie, de nombreux travaux ont étudié les corrélations dans lARNm et données dexpression des protéines (voir ci-dessous et tableau 1). En théorie, lorsque deux échantillons contenant des données de haute dimension (comme les microréseaux et la protéomique) sont comparés, les analyses de corrélation fournissent une mesure de lécart par rapport à lunité comme source de différence entre les échantillons . En bref, deux échantillons avec des informations identiques et complètement non identiques afficheront respectivement une corrélation unitaire (R2 = 1) et nulle (R2 = 0).
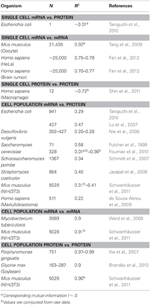
Tableau 1. ARNm et protéine expr corrélations de session dans divers organismes.
La corrélation parfaite (R2 = 1) est une situation idéalisée qui est loin de la réalité, car technique ou le bruit expérimental seul interfère et réduit la corrélation. De plus, les dernières années ont mis en évidence lexistence dun bruit biologique: les études sur des cellules et molécules individuelles ont montré une stochasticité dans la dynamique dexpression génique due à leffet combinatoire des faibles nombres de copies moléculaires et à la nature quantique de la dynamique des promoteurs (Raj et van Oudenaarden, 2009; Eldar et Elowitz, 2010). Dautre part, les populations clonales de cellules présentent une hétérogénéité des niveaux dune expression protéique donnée par cellule à tout moment mesuré (Chang et al., 2008). Ensemble, la stochasticité et lhétérogénéité sont essentielles pour produire la diversification du destin cellulaire, les variations phénotypiques et lamplification des signaux intracellulaires (Locke et al., 2011; Selvarajoo, 2012).
Les fluctuations stochastiques, ou bruit intrinsèque, provoquent lexpression dune espèce moléculaire varie dans le temps et entre les cellules, conduisant à des réponses non corrélées (Elowitz et al., 2002). Ceci est particulièrement important pour les ARNm et les protéines à faible nombre de copies. Ainsi, la corrélation entre les échantillons (cellules) peut être diminuée en raison du bruit intrinsèque (figure 1A). Dautres sources de bruit biologique dues à des facteurs extrinsèques comprennent la variabilité de la taille des cellules, le nombre de copies moléculaires et les fluctuations environnementales entre les cellules individuelles.Ces facteurs déforment le dogme central déterministe et modifient probablement les corrélations fortes en corrélations plus faibles (Figure 1B).
Une étude récente a comparé les expressions dARNm et de protéines dEscherichia coli entre des cellules individuelles au niveau dune seule molécule et a fourni un scénario qui remet en question profondément le dogme central. Taniguchi et coll. (2010) ont révélé quil ny a pas de corrélation (R2 ~ 0) entre lARNm de tufA individuel et les niveaux de protéines dans des cellules individuelles. Notamment, ils ont conclu que le manque de corrélation est probablement dû à des différences dans la durée de vie de lARNm et des protéines. Bien que ce soit une explication plausible, Taniguchi et al. ont pris soin de ne pas réfuter lhypothèse de longue date en affirmant que les moyennes temporelles des niveaux dARNm devraient être en corrélation avec les niveaux de protéines. Cependant, aucune preuve na montré que cétait le cas réel, et lorsque nous avons évalué les dépendances non linéaires en utilisant linformation mutuelle (Steuer et al., 2002; Tsuchiya et al., 2010) dans Taniguchi et al. ensemble de données, nous avons trouvé le résultat non-dépendant, cest-à-dire I ~ 0. Cela confirme que les expressions ARNm à protéine entre les cellules individuelles au niveau dune seule molécule sont clairement sans rapport. De plus, lors dun zoom au niveau dune seule molécule dans le diagramme de corrélation, il est évident que leurs corrélations par paires sont faibles (Figure 1A, insérer, à titre dillustration).
Notamment, au niveau de la population cellulaire, Taniguchi et Al. ont pu montrer une corrélation relativement élevée entre les expressions de lARNm et des protéines avec R2 = 0,29 (figure 2A). En fait, une autre étude indépendante de Lu et al. (2007), pour la population dE. Coli, ont également montré une corrélation relativement élevée (R2 = 0,47). Des analyses similaires effectuées sur Saccharomyces cerevisiae (Futcher et al., 1999), fibroblaste murin NIH / 3T3 (Schwanhäusser et al., 2011) et plusieurs autres populations cellulaires (Nie et al., 2006; Schmidt et al., 2007; Jayapal et al., 2008; de Sousa Abreu et al., 2009) ont tous montré des structures corrélées entre les expressions à léchelle du transcriptome et à léchelle du protéome (tableau 1). Alors, pourquoi ny a-t-il pas de corrélation entre les expressions dARNm et de protéines individuelles dans des cellules individuelles, alors quau niveau de la population, des relations collectives sont observées entre les expressions dARNm et de protéines à grande échelle?
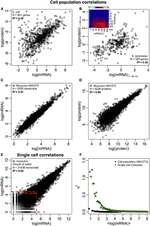
Figure 2. Corrélations dexpressions à léchelle Omics. Populations cellulaires: corrélations ARNm-protéines chez (A) E. coli (Taniguchi et al., 2010) et (B) S. cerevisiae (Fournier et al., 2010) entre les expressions dARNm à t = 60 min et les expressions protéiques à t = 360 min. Insertion: la matrice de corrélation entre tous les points temporels montre une augmentation retardée des corrélations entre lARNm et les protéines. (C) expressions de lARNm et de la protéine (D) entre deux échantillons de cellules NIH / 3T3 murines (Schwanhäusser et al., 2011). Cellules uniques: expressions dARNm (E) entre deux ovocytes (Tang et al., 2009). Les lignes pointillées rouges indiquent les régions dexpressions dARNm faibles (log (ARNm) < 5). (F) Bruit (η2) versus log (expressions de lARNm) pour la population cellulaire (NIH / 3T3, points noirs, Schwanhäusser et al., 2011) et les cellules individuelles (ovocytes, triangles verts, Tang et al., 2009). Chaque point représente la valeur dun groupe de P = 100 ARNm. η2 est proche de zéro pour la population cellulaire pour toutes les expressions dARNm. Pour les cellules individuelles, η2 est le plus élevé pour les ARNm avec les nombres de copies les plus bas et sapproche de zéro pour les nombres de copies plus élevés.
Nous pensons il y a deux raisons majeures aux différences. Premièrement, comme indiqué précédemment, le bruit, quil soit de nature biologique ou non biologique, réduit la corrélation. Puisque les analyses sur des cellules individuelles ont montré limportance de la stochasticité et de la variabilité, ces effets sont cruciaux pour réduire les corrélations de cellule unique. Au niveau de lensemble, lorsque les cellules sont échantillonnées dans une population, le bruit total (intrinsèque + extrinsèque) est réduit, car le bruit aléatoire sannule sur toute la gamme dexpressions moléculaires (figures 1C-F), pour révéler la réponse moyenne et lauto-organisation ( Karsenti, 2008; Selvarajoo, 2011; Hekstra et Leibler, 2012; Selvarajoo et Giuliani, 2012). Par conséquent, un bon degré de corrélation dexpression ARNm-protéine émerge. Deuxièmement, pour létude sur une seule cellule (Taniguchi et al., 2010), la corrélation dexpression ARNm-protéine individuelle a été comparée à travers de nombreuses cellules. Dans les études de population cellulaire, cependant, la comparaison est faite dans son intégralité, à travers des milliers dARNm et de protéines sur plusieurs ordres de grandeur supérieurs à la plage dexpression trouvée pour une seule molécule entre les cellules. Cela conduit donc à des corrélations plus élevées au niveau de la population, car leffet des variations moléculaires uniques devient négligeable.
Malgré les structures corrélées observées pour les populations de cellules, il existe des raisons tangibles à lécart important par rapport à la corrélation parfaite.Comme indiqué précédemment, un point clé est que les ARNm et les protéines sont séquentiellement localisés avec plusieurs processus manquants, non représentés dans le dogme central. Lajout des intermédiaires manquants le long dune voie biochimique entraînera un retard notable dans le flux dinformations (Selvarajoo, 2006, 2011; Piras et al., 2011), et la corrélation entre eux pourrait en souffrir. Cela pourrait également faire partie du fait noté par Taniguchi et al. que les expressions de lARNm et des protéines ont des durées de vie différentes. Notamment, cette postulation est étayée par un travail récent sur S. cerevisiae traité avec la rapamycine qui a montré que les corrélations temporelles de lexpression de lARNm-protéine étaient initialement faibles, R2 = 0,01 à 40 min, néanmoins, sur 360 min après la perturbation, la corrélation a augmenté, R2 = 0,36 (Fournier et al., 2010, figure 2B). Les données indiquent quen cas de perturbation chimique, la réponse initiale entre les expressions de lARNm et des protéines dévie en raison du retard et des différents mécanismes cinétiques entre eux, ainsi que des effets secondaires tels que linterférence de signalisation autocrine ou paracrine (Shvartsman et al., 2002; Isalan et al., 2008). Lorsque les effets de la perturbation sont atténués au fil du temps, la récupération des corrélations sest produite.
Pour vérifier davantage lhypothèse selon laquelle les processus de retard séquentiel ou différentes durées de vie sont cruciales pour diminuer les corrélations ARNm-protéine, nous avons comparé R2 entre les même espèce moléculaire du dogme central (par exemple, entre lARNm et lARNm) dans les populations cellulaires et les cellules individuelles. La corrélation dexpression de lARNm-ARNm à léchelle du transcriptome entre les réplicats déchantillons de population de cellules NIH / 3T3 (Schwanhäusser et al., 2011) (Figure 2C) et Mycobacterium tuberculosis (Ward et al., 2008) est très élevée, avec R2 > 0.9 (Tableau 1). De telles corrélations fortes sont également observées entre les échantillons de population pour les expressions protéine – protéine dans les cellules NIH / 3T3 (Schwanhäusser et al., 2011) (Figure 2D), Porphyromonas gingivalis (Xia et al., 2007) et Glycine max (Brandão et al. , 2010) (Tableau 1). Étant donné que ces données qui comparent les mêmes espèces produisent des corrélations très élevées, il est concevable que les processus de retard séquentiel ou différentes durées de vie soient responsables de labaissement des structures de corrélation au niveau de la population entre les expressions de lARNm et des protéines.
Dans les ovocytes murins simples ( Tang et al., 2009), lors de la comparaison des expressions ARNm-ARNm entières, une structure hautement corrélée est observée (R2 = 0,92, figure 2E). Cependant, en se concentrant uniquement sur les ARNm faiblement exprimés (avec des expressions logarithmiques < 5), le bruit stochastique abaisse considérablement la corrélation par paire (R2 < 0,54). Pour sonder ce résultat, nous avons évalué le bruit, η2 = σ2XY / μ2XY, à travers des expressions dARNm entières (figure 2F). Nous avons noté que η2 est le plus élevé pour les expressions les plus basses, en raison de leffet prononcé des fluctuations stochastiques par rapport à leurs expressions, et se rapproche de zéro pour les expressions plus élevées, où ce bruit devient moins significatif (Piras et al., 2012). Pour la population cellulaire, comme prévu, un bruit proche de zéro est observé sur toute la plage dexpression en raison de lannulation du bruit aléatoire (figures 1E, F).
Des structures hautement corrélées pour des expressions entières dARNm – ARNm étaient également rapportés pour une seule cellule cancéreuse (Fan et al., 2012), bien que moins significatif avec R2 ~ 0,7 (tableau 1). En outre, la comparaison des expressions protéine-protéine dans les macrophages humains stimulés par le LPS a également montré des corrélations élevées, R2 ~ 0,72 (Shin et al., 2011) (tableau 1). Bien quil ny ait pas de corrélation entre les expressions individuelles dARNm-protéines dans des cellules individuelles, la corrélation à grande échelle ou à léchelle de lomique entre les mêmes espèces moléculaires dans des cellules individuelles est très élevée.
Ainsi, quil sagisse de cellules uniques ou de populations de cellules , les données à léchelle de lomique indiquent que les corrélations entre les mêmes espèces moléculaires (ARNm vs ARNm et protéine vs protéine) sont sensiblement plus élevées quentre différentes espèces (ARNm vs protéine). Cela reflète le fait que bien que les processus de retard et les durées de vie différentes soient essentiels pour réduire les corrélations, ces mécanismes ne sont pas suffisants pour supporter le manque de structure de corrélation observée entre les transcriptions individuelles des cellules individuelles vers les expressions protéiques.
Donc loin, en étudiant les expressions à grande échelle des ARNm et des protéines de divers systèmes cellulaires, nous avons montré que les structures de corrélation émergent à léchelle mondiale. Cependant, les analyses de corrélation ne révèlent que la connectivité entre deux échantillons testés, et ne montrent pas la direction de flux dinformations. Pour que le dogme central soit valable à léchelle mondiale, le flux global dinformations doit aller de lADN aux protéines. Ce flux dinformations a été démontré par une myriade dautres études qui impliquent de perturber les récepteurs des populations cellulaires et de surveiller les dynamique des facteurs de transcription se liant à lADN et linduction dexpressions géniques à grande échelle (figure 3A).Par exemple, dans le cas des cellules immunitaires stimulées par le LPS, il a été démontré que lactivation du facteur de transcription NF-κB se produit vers 15 min (Liu et al., 1999), linduction de ses gènes en aval vers 30 min (Liu et al., 1999; Xaus et al., 2000; Selvarajoo et al., 2008), et la traduction des protéines correspondantes dans la région de 60 à 90 min (Kawai et al., 1999; Xaus et al. ., 2000) (Figure 3B). Une telle direction séquentielle de la transcription globale vers le flux dinformations de traduction est également observée pour les systèmes bactériens, comme E. coli, au niveau de la population cellulaire (Golding et al., 2005).

Figure 3. Le flux dinformation du dogme central. (A) Schéma de lexpression de TNF induite par LPS / TLR4, via le facteur de transcription NF-κB et le gène tnf, suivant un flux dinformation linéaire. (B) Profils temporels expérimentaux de lactivité de liaison du promoteur des expressions NF-κB (panneaux supérieurs), tnf (panneaux du milieu) et TNF (panneaux inférieurs) au niveau de la population cellulaire. (C) Profils temporels schématiques de la dynamique du promoteur, de lARNm et des expressions protéiques au niveau de la cellule unique (Raj et van Oudenaarden, 2009).
Alternativement, les investigations à la résolution dune seule cellule révèlent des fluctuations aléatoires sur le flux dinformation linéaire: les facteurs de transcription se liant aux régions promotrices de lADN sont quantiques, entraînant un comportement déclatement de la transcription de lARNm et, par la suite, induisent une variabilité dans la traduction des protéines, même entre des cellules identiques (figure 3C) (Raj et van Oudenaarden, 2009; Eldar et Elowitz, 2010; Locke et al., 2011; Hekstra et Leibler, 2012; Selvarajoo, 2012). En conséquence, à un moment donné, la réponse moléculaire individuelle pour des cellules individuelles est plutôt bruyante par rapport à léchelle moyenne de la population (Selvarajoo, 2011).
Conclusions
Les exemples présentés dans cet article met en évidence les différences dans lordre des valeurs de corrélation observées entre les espèces dans le dogme central sur les populations cellulaires et les cellules individuelles. Les analyses statistiques des populations cellulaires brossent un tableau selon lequel la corrélation dexpression entre la même espèce moléculaire est très élevée et entre les espèces est modérément élevée. Bien que les corrélations cellulaires entre les mêmes espèces soient comparables avec les populations cellulaires, elles ont montré une plus grande dispersion dans leurs parcelles dexpressions en raison de leffet prononcé du bruit biologique, en particulier pour les transcriptions avec un faible nombre de copies. Notamment, la corrélation par paire de cellules individuelles «devient nulle pour les molécules individuelles (Taniguchi et al., 2010). En fait, les fluctuations stochastiques et la variabilité des expressions moléculaires sont connues pour être fonctionnelles dans la prise de décision sur le sort des cellules et le basculement des états cellulaires (Losick et Desplan, 2008; Eldar et Elowitz, 2010; Kuwahara et Schwartz, 2012). , 2008) résultant en des réponses moyennes émergentes. En analysant un petit nombre ou des molécules individuelles, la structure de corrélation ne peut pas être observée.
Dans lensemble, il est concevable que la visualisation du flux dinformations dun ADN unique vers une protéine remette en question le dogme central car la réponse de chaque molécule à un moment donné ne sera probablement pas corrélée. Cependant, globalement, lobservation de la réponse déterministe moyenne suggère que léquilibre net de la genette les informations restent à lextrême droite des sentiers. Par conséquent, le dogme central doit être considéré comme un flux dinformation cellulaire macroscopique à léchelle omique, et non au niveau dun gène à une protéine. En tant que tel, nous pensons que sa simplicité restera lun des piliers théoriques les plus influents des systèmes vivants.
Déclaration de conflit dintérêts
Les auteurs déclarent que la recherche a été menée en labsence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit dintérêts potentiel.
Remerciements
Kentaro Hayashi est remercié pour ses commentaires. Le fonds de recherche de la ville de Tsuruoka et de la préfecture de Yamagata est apprécié pour son soutien.
Crick, F. (1958). Sur la synthèse des protéines. Symp. Soc. Exp. Biol. 12, 139-163.
Résumé de Pubmed | Texte intégral de Pubmed
Crick, F. (1970). Dogme central de la biologie moléculaire. Nature 227, 561–563.
Pubmed Abstract | Texte intégral de Pubmed
Hayden, E. C. (2011). La preuve de lARN modifié suscite le débat. Nature 473, 432.
Résumé de Pubmed | Pubmed Texte intégral | CrossRef Full Text
Hekstra, D. R. et Leibler, S. (2012). Contingence et lois statistiques dans des écosystèmes microbiens fermés répliqués. Cell 149, 1164–1173.
Résumé de Pubmed | Pubmed Texte intégral | CrossRef Full Text
Kuwahara, H., et Schwartz, R. (2012).Gain à létat déquilibre stochastique dans un processus dexpression génique avec contrôle de la dégradation de lARNm. J. R. Soc. Interface 9, 1589–1598.
Résumé de Pubmed | Pubmed Texte intégral | CrossRef Full Text
Nie, L., Wu, G. et Zhang, W. (2006). Corrélation de lexpression de lARNm et labondance des protéines affectées par de multiples caractéristiques de séquence liées à lefficacité de la traduction chez Desulfovibrio vulgaris: une analyse quantitative. Genetics 174, 2229–2243.
Pubmed Abstract | Pubmed Texte intégral | CrossRef Texte intégral
Prusiner, S. B. (1998). Prions. Proc. Natl. Acad. Sci. U.S.A. 95, 13363–13383.
Pubmed Abstract | Pubmed Texte intégral | Texte intégral CrossRef
Rosner, B. (2011). Fondamentaux de la biostatistique. 7e éd. Boston, MA: Duxbury Press.
Selvarajoo, K. (2006). Découverte des mécanismes dactivation différentielle des voies de signalisation du récepteur de type péage 4 dans les knockouts MyD88. FEBS Lett. 580, 1457–1464.
Résumé de Pubmed | Pubmed Texte intégral | Texte intégral CrossRef
Selvarajoo, K. (2011). Loi macroscopique de conservation révélée dans la dynamique de population de la signalisation des récepteurs de type péage. Cell Commun. Signal. 9, 9.
Résumé de Pubmed | Pubmed Texte intégral | Texte intégral CrossRef
Selvarajoo, K. (2012). Comprendre les décisions biologiques multimodales à partir dune seule cellule et de la dynamique des populations. Wiley Interdiscip. Rev. Syst. Biol. Med. 4, 385–399.
Résumé de Pubmed | Pubmed Texte intégral | CrossRef Texte intégral
Stewart, T. R. (1990). Une décomposition du coefficient de corrélation et son utilisation dans lanalyse des compétences de prévision. Prévisions météorologiques. 5, 661–666.